Aux “Rencontres R 2018” à Rennes, j’ai proposé une brève introduction à la cartographie en utilisant le récent package {sf} et quelques autres packages intéressant. Ce post de blog est une version enrichie de ma présentation et me permet de partager le code des différentes cartes qui y figurent. Cela reste une brève introduction.
La présentation originale des RR2018 est sur mon dépôt github
Le code R de cet article est caché, vous pouvez le faire apparaître en cliquant sur les boutons “CODE”
La cartographie dans R
Toutes les opérations classiques sur des données spatialisées peuvent être totalement réalisées dans :
- Lecture et exploration des données spatialisées / géographiques
- Manipulation des attributs (création, sélection)
- Traitements géomatiques (intersection, jointure, calcul de surface)
- Création de cartes (statiques, interactives)
Packages
library(dplyr)
library(sf)
library(ggplot2)
library(tmap)
library(leaflet)Les projections : le ❤️ du problème
Une carte est une représentation en 2 dimensions de tout ou partie de notre planète qui, elle, désolé de vous l’apprendre, est une sphère !
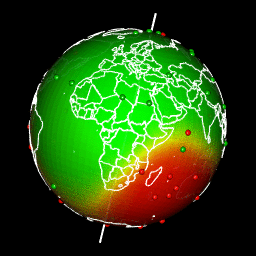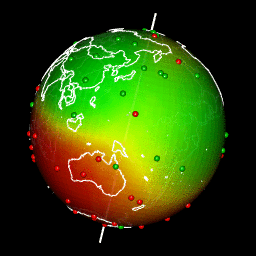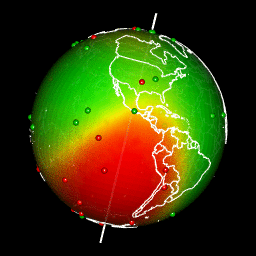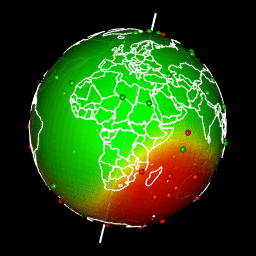
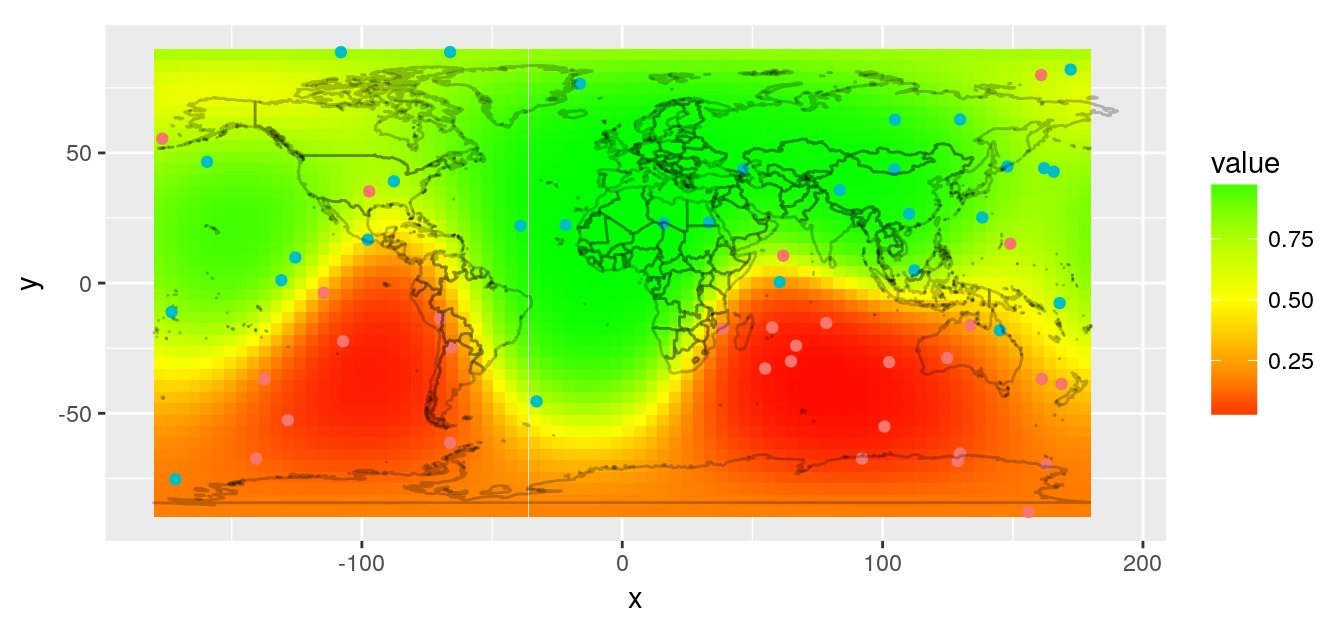
Images de l’article “interpolation spatiale sur le globe terrestre 3D”
Pour créer une carte en 2 dimensions, il faut réaliser une projection. Les zones que vous étudiez seront plus ou moins déformées par la projection que vous aurez choisie. Un jour ou l’autre (et ça arrivera plus vite que vous ne le pensez !), vous aurez un problème avec une projection. Si vous ne savez pas ce qu’est une projection, vous ne trouverez pas l’origine de votre problème ! Donc, si vous voulez faire de la cartographie avec R sans passer par une formation (telle qu’une formation chez ThinkR par exemple), vous allez devoir apprendre et comprendre ce qu’est une projection.
=> Retenez votre première fonction sf::st_transform(x, crs) pour changer de système de coordonnées
Projection de la France Métropolitaine
Je ne vais pas vous faire un cours sur les projections. Ce serait très utile, mais ce n’est pas le coeur de cet article. Je vous présente juste la carte de la France Métropolitaine que vous pouvez télécharger sur mon github, mais ce sont des données récupérées sur le site web de l’ign.
Télécharger les données
# extraWD <- "."
if (!file.exists(file.path(extraWD, "departement.zip"))) {
githubURL <- "https://github.com/statnmap/blog_tips/raw/master/2018-07-14-introduction-to-mapping-with-sf-and-co/data/departement.zip"
download.file(githubURL, file.path(extraWD, "departement.zip"))
unzip(file.path(extraWD, "departement.zip"), exdir = extraWD)
}
departements_L93 <- st_read(dsn = extraWD, layer = "DEPARTEMENT",
quiet = TRUE) %>%
st_transform(2154)- 🌍 Coordonnées géographiques
- En degrés, minutes
- EPSG: 4326
- Utilisation pour partager les données
ggplot(departements_L93) +
aes(fill = CODE_REG) +
scale_fill_viridis_d() +
geom_sf() +
coord_sf(crs = 4326) +
guides(fill = FALSE) +
ggtitle("Coord. géographiques") +
theme(title = element_text(size = 16),
plot.margin = unit(c(0,0.1,0,0.25), "inches"))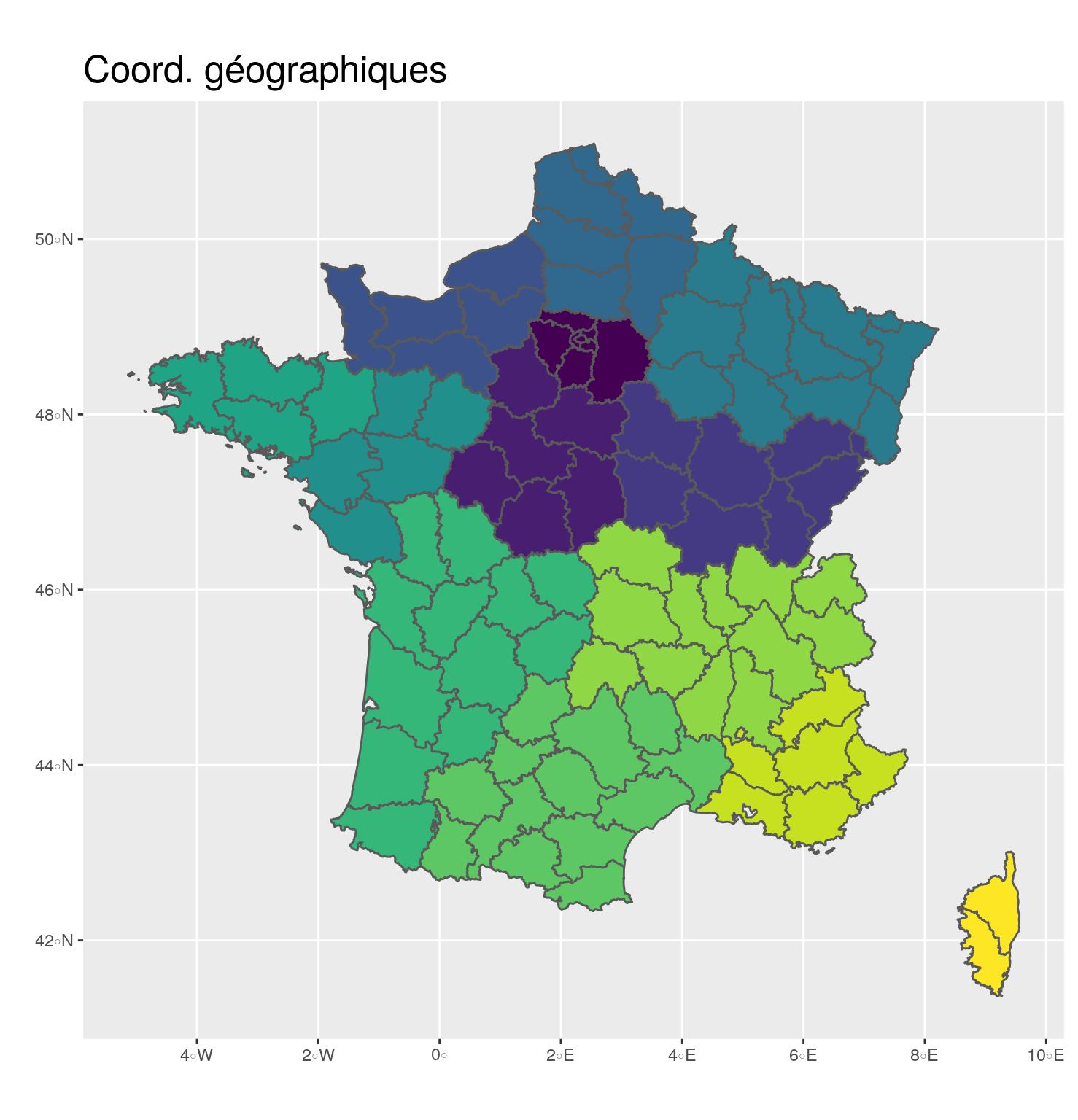
- 🗺 Projection Lambert 93
- Coordonnées en mètres
- EPSG: 2154
- Une projection pour afficher une carte de France Métropolitaine
g_dept <- ggplot(departements_L93) +
aes(fill = CODE_REG) +
scale_fill_viridis_d() +
geom_sf() +
coord_sf(crs = 2154, datum = sf::st_crs(2154)) +
guides(fill = FALSE) +
ggtitle("Lambert 93") +
theme(title = element_text(size = 16))
g_dept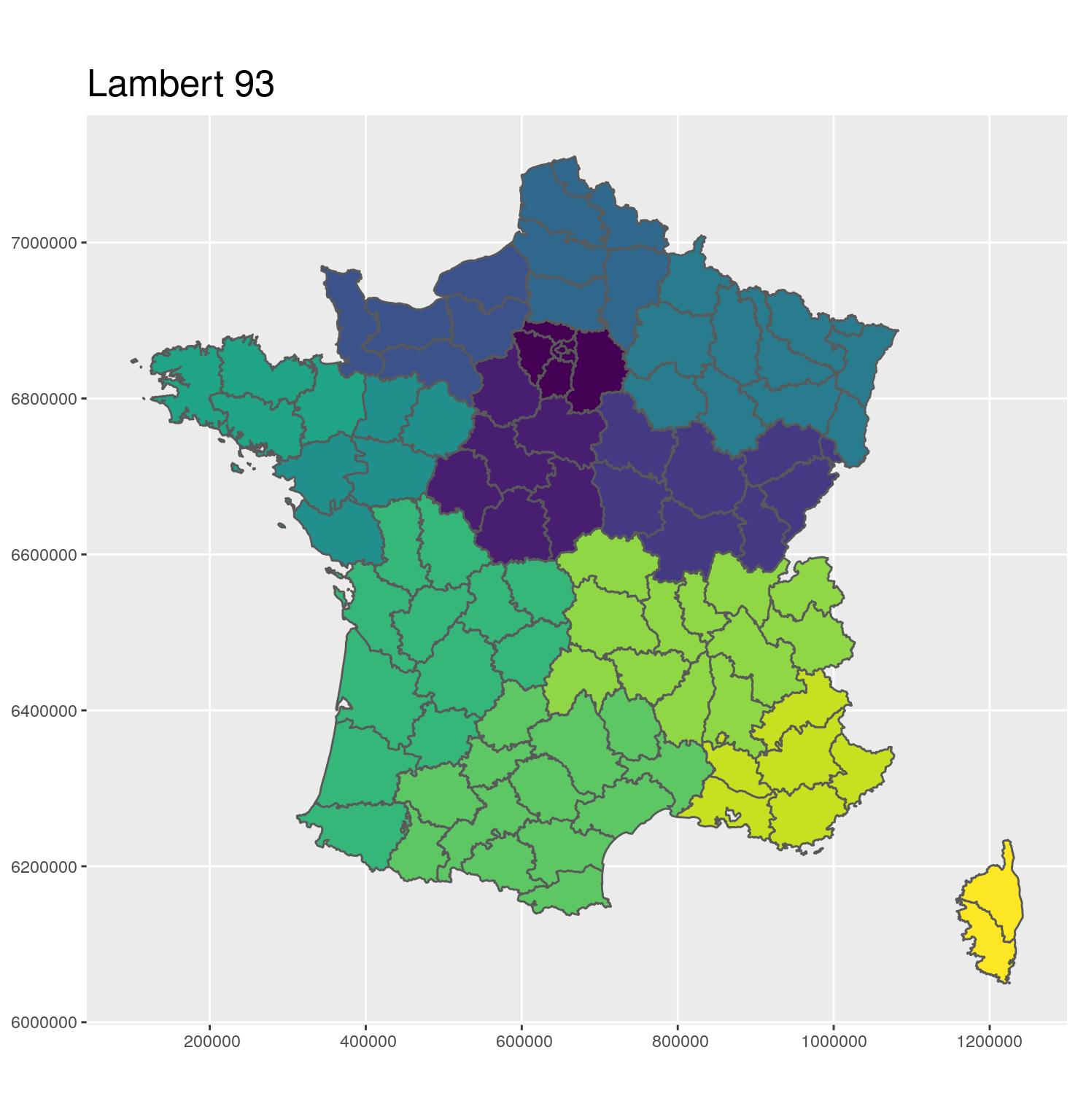
Un GIF pour mieux voir la différence
library(magick)
newlogo <- image_scale(image_read(glue("../../static{StaticImgWD}/frwgs84-1.jpeg")), "x1000")
oldlogo <- image_scale(image_read(glue("../../static{StaticImgWD}/frl93-1.jpeg")), "x1000")
image_animate(c(oldlogo, newlogo), fps = 1, loop = 0)
La cartographie et les analyses spatiales avec R
Nous allons nous focaliser uniquement sur les données vectorielles. La manipulation des données raster se fait avec le package {raster}, qui est très puissant, mais ce n’est pas le sujet de ce post.

Format des fichiers de couches vectorielles
- Avec {sf} : lecture avec
st_read- Tous les formats géré par GDAL (http://www.gdal.org/)
- CSV, Mapinfo, Google Earth, GeoJSON, PostGIS, …
- Format shapefile de ESRI™️
- Le plus couramment partagé
- Attention : 4 fichiers minimum (shp, shx, dbf, prj)
- Tous les formats géré par GDAL (http://www.gdal.org/)
Avec {sf}, les fichiers deviennent des tables de données classiques.
departements_L93[,2:3]## Simple feature collection with 96 features and 2 fields
## geometry type: MULTIPOLYGON
## dimension: XY
## bbox: xmin: 99217.1 ymin: 6049646 xmax: 1242417 ymax: 7110480
## epsg (SRID): 2154
## proj4string: +proj=lcc +lat_1=49 +lat_2=44 +lat_0=46.5 +lon_0=3 +x_0=700000 +y_0=6600000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs
## First 10 features:
## CODE_DEPT NOM_DEPT geometry
## 1 39 JURA MULTIPOLYGON (((886244.2 66...
## 2 42 LOIRE MULTIPOLYGON (((764370.3 65...
## 3 76 SEINE-MARITIME MULTIPOLYGON (((511688.8 69...
## 4 89 YONNE MULTIPOLYGON (((709449.1 67...
## 5 68 HAUT-RHIN MULTIPOLYGON (((992779.1 67...
## 6 28 EURE-ET-LOIR MULTIPOLYGON (((548948.9 68...
## 7 10 AUBE MULTIPOLYGON (((740396.5 68...
## 8 55 MEUSE MULTIPOLYGON (((846578.7 68...
## 9 61 ORNE MULTIPOLYGON (((425026.6 68...
## 10 67 BAS-RHIN MULTIPOLYGON (((998020.8 68...Contrairement à {sp} + {rgdal}
str(as(departements_L93[,2:3], "Spatial"), max.level = 2)## Formal class 'SpatialPolygonsDataFrame' [package "sp"] with 5 slots
## ..@ data :'data.frame': 96 obs. of 2 variables:
## ..@ polygons :List of 96
## ..@ plotOrder : int [1:96] 57 12 27 69 76 21 63 18 65 26 ...
## ..@ bbox : num [1:2, 1:2] 99217 6049646 1242417 7110480
## .. ..- attr(*, "dimnames")=List of 2
## ..@ proj4string:Formal class 'CRS' [package "sp"] with 1 slot# str(as(departements_L93[,2:3], "Spatial"), max.level = 3)Manipulation des données avec {dplyr}
- 🎉 Tout ce que vous connaissez de {dplyr} fonctionne sur les objets de {sf} 🎉
%>%select,mutatepour les attributs (= colonnes)filter,arrangepour les entités (= lignes)
Exemple de manipulation
Bret_L93 <-
departements_L93 %>%
mutate_at(
vars(NOM_DEPT, NOM_REG),
tolower) %>%
select(CODE_DEPT, NOM_DEPT, NOM_REG) %>%
filter(NOM_REG == "bretagne")
Bret_L93## Simple feature collection with 4 features and 3 fields
## geometry type: MULTIPOLYGON
## dimension: XY
## bbox: xmin: 99217.1 ymin: 6704195 xmax: 400572.3 ymax: 6881964
## epsg (SRID): 2154
## proj4string: +proj=lcc +lat_1=49 +lat_2=44 +lat_0=46.5 +lon_0=3 +x_0=700000 +y_0=6600000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs
## CODE_DEPT NOM_DEPT NOM_REG geometry
## 1 35 ille-et-vilaine bretagne MULTIPOLYGON (((330478.6 68...
## 2 22 cotes-d'armor bretagne MULTIPOLYGON (((259840 6876...
## 3 29 finistere bretagne MULTIPOLYGON (((116158.5 67...
## 4 56 morbihan bretagne MULTIPOLYGON (((256230.1 67...ggplot(Bret_L93) +
geom_sf(aes(fill = NOM_DEPT)) +
scale_fill_viridis_d()
- Fusion d’entités avec
group_by+summarize
Avant
g_dept +
ggtitle("Départements")
Fusion
region_L93 <- departements_L93 %>%
group_by(CODE_REG, NOM_REG) %>%
summarize()Après
g_region <- ggplot(region_L93) +
aes(fill = CODE_REG) +
scale_fill_viridis_d() +
geom_sf() +
coord_sf(crs = 2154, datum = sf::st_crs(2154)) +
guides(fill = FALSE) +
ggtitle("Régions") +
theme(title = element_text(size = 16))
g_region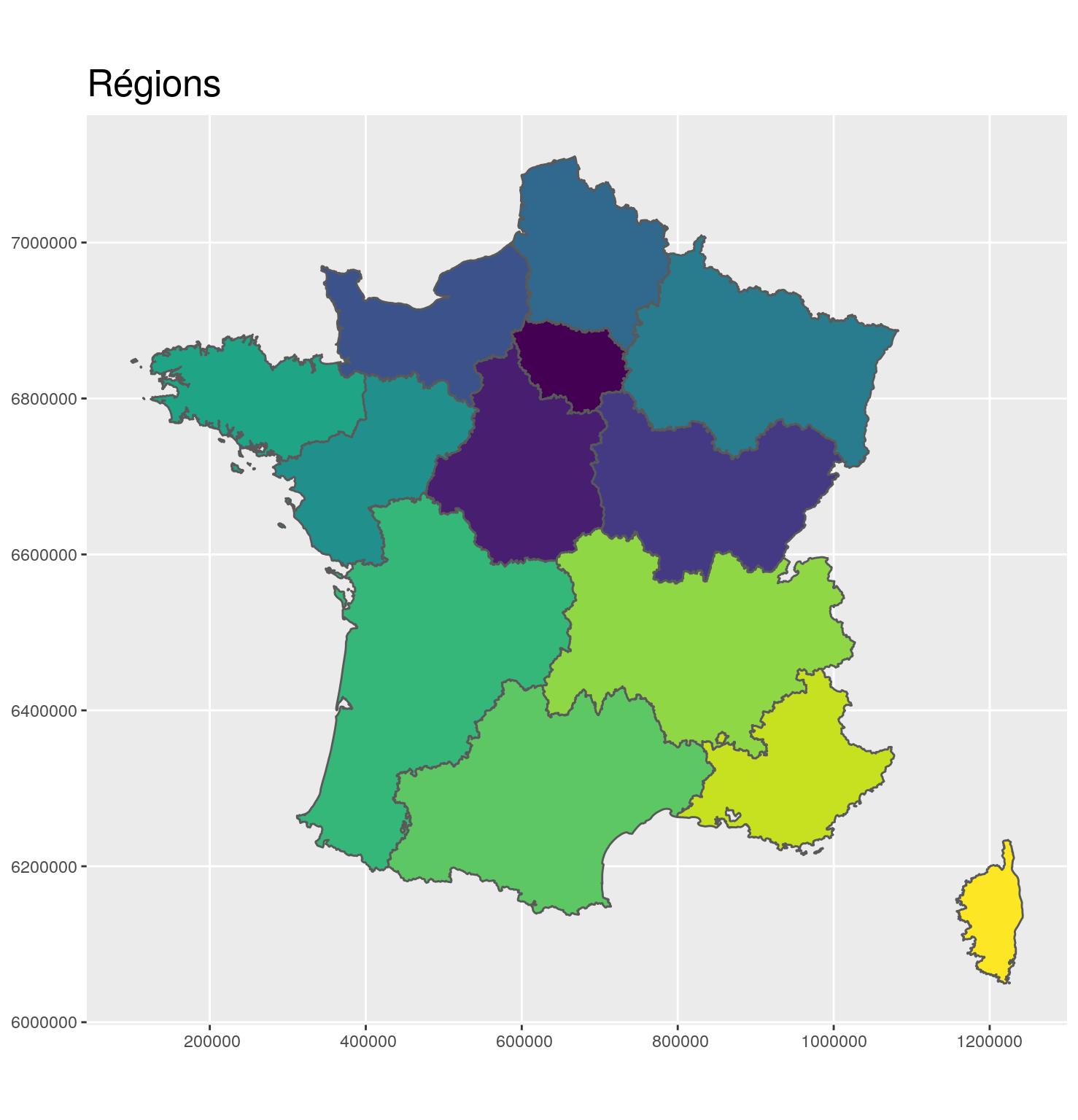
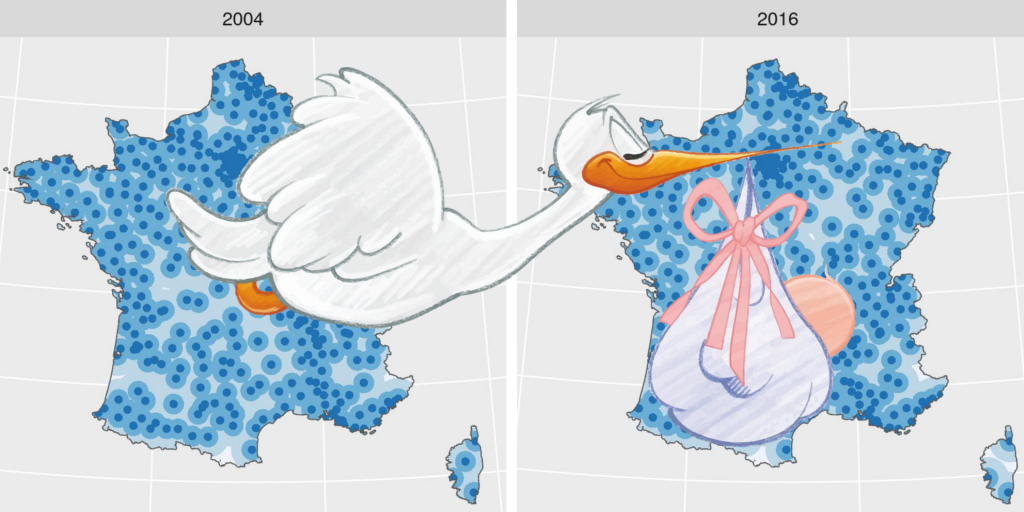
- Jointure classique entre un shapefile des communes de France et un fichier CSV de communes française ayant une maternité avec
left_join- La carte de communes est issue du site web de l’ign. Vous pouvez la télécharger sur mon github aussi.
- Les données de maternités viennent du portail open-data du ministère des solidarités et de la santé. Ce sont les bases “SAE - La statistique annuelle des établissements de santé”. Sur mon github ici, un fichier Rmd décrit la façon dont j’ai préparé le jeu de données de maternités.
Télécharger les données
# extraWD <- "."
# -- Communes --
if (!file.exists(file.path(extraWD, "communes.zip"))) {
githubURL <- "https://github.com/statnmap/blog_tips/raw/master/2018-07-14-introduction-to-mapping-with-sf-and-co/data/communes.zip"
download.file(githubURL, file.path(extraWD, "communes.zip"))
}
unzip(file.path(extraWD, "communes.zip"), exdir = extraWD)
# -- Maternites --
if (!file.exists(file.path(extraWD, "communes.zip"))) {
githubURL <- "https://github.com/statnmap/blog_tips/raw/master/2018-07-14-introduction-to-mapping-with-sf-and-co/data-mater/Maternite_2004-2016.csv"
download.file(githubURL, file.path(extraWD, "Maternite_2004-2016.csv"))
}
# Read shapefile of French communes
communes <- st_read(dsn = extraWD, layer = 'COMMUNE', quiet = TRUE) %>%
select(NOM_COM, INSEE_COM)
# Read file of maternities for 2016
data.maternite <- readr::read_csv(file.path(extraWD, "Maternite_2004-2016.csv")) %>%
filter(an == 2016)Jointure
# Join database with shapefile by attributes
maternites_L93 <- communes %>%
right_join(data.maternite,
by = "INSEE_COM") %>%
st_transform(2154) g_region +
geom_sf(data = maternites_L93, fill = "white", colour = "white") +
coord_sf(crs = 2154, datum = sf::st_crs(2154)) +
ggtitle("Communes avec une maternité en 2016")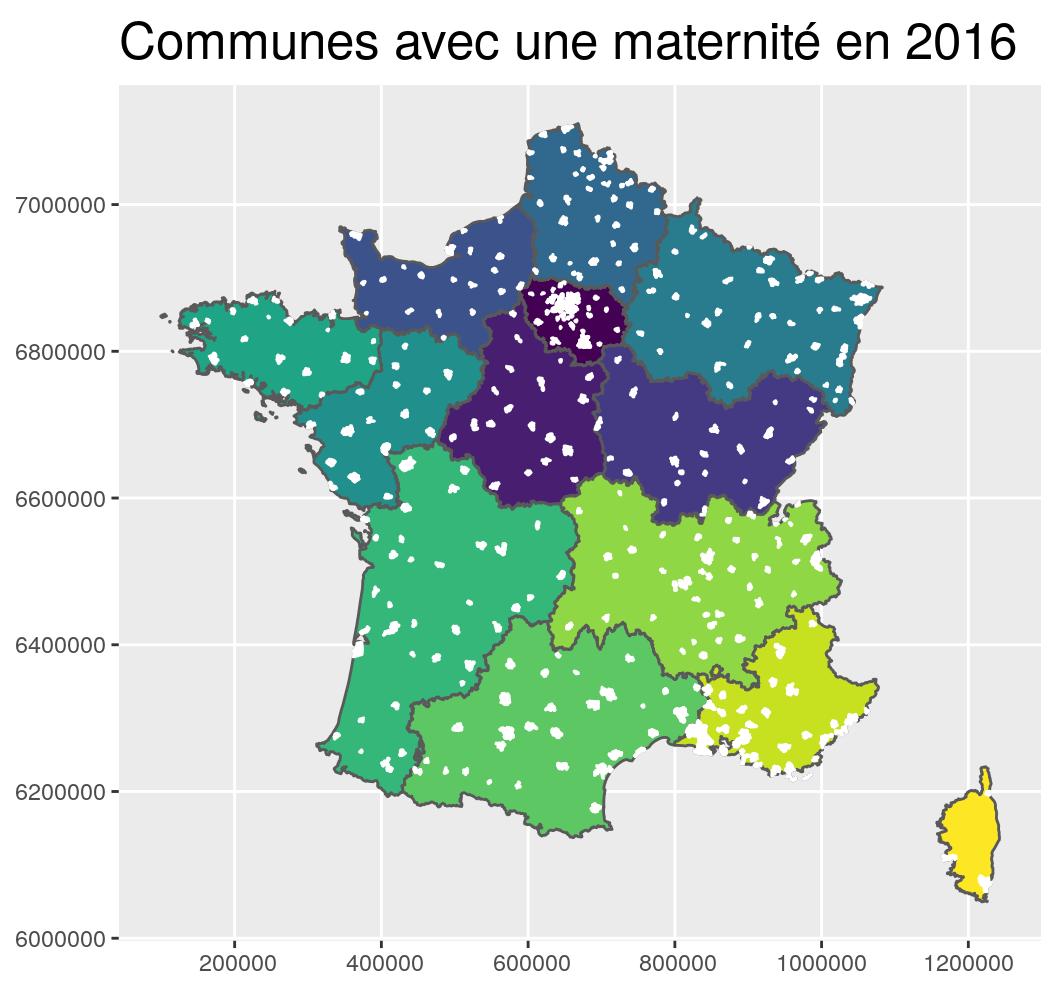
Jointures spatiales
Joindre des couches avec st_intersection pour sélectionner spatialement et récupérer les informations d’une autre couche spatiale en fonction des positions géographiques.
- Points, polygones, lignes avec polygones
Jointure spatiale
maternites_Bret_L93 <- maternites_L93 %>%
st_intersection(Bret_L93)## Attributs du fichier 'Maternités' avant intersection## [1] "NOM_COM" "INSEE_COM" "an" "Code_Postal" "n"
## [6] "geometry"## Attributs après Intersection avec région## [1] "NOM_COM" "INSEE_COM" "an" "Code_Postal" "n"
## [6] "CODE_DEPT" "NOM_DEPT" "NOM_REG" "geometry"ggplot() +
geom_sf(data = departements_L93, colour = "grey20") +
geom_sf(data = Bret_L93, fill = "#440154", colour = "grey60") +
geom_sf(data = maternites_Bret_L93,
fill = "#FDE725", colour = "#FDE725") +
coord_sf(crs = 2154, datum = sf::st_crs(2154),
xlim = st_bbox(Bret_L93)[c(1,3)],
ylim = st_bbox(Bret_L93)[c(2,4)]
) +
theme(title = element_text(size = 16),
panel.background = element_rect(fill = "#66BDFF")) +
ggtitle("Communes bretonnes avec maternité")
Opérations de géomatique
- Calcul de zone tampon pour mettre en évidence la région Bretagne avec
st_buffer
Zone tampon
# Buffer area for Brittany
Bret_buffer10_L93 <-
Bret_L93 %>%
st_buffer(
dist =
units::set_units(10, km)
) %>%
st_cast() # useful sometimes !
# Buffer Bretagne for larger study area bbox only
Bret_buffer30_L93 <-
Bret_L93 %>%
st_buffer(
dist =
units::set_units(30, km)
) ggplot() +
geom_sf(data = departements_L93, colour = "grey50",
fill = "white") +
geom_sf(data = st_union(Bret_buffer10_L93), fill = "#FDE725", #"#440154",
colour = "grey80", size = 1.1) +
geom_sf(data = st_union(departements_L93), colour = "grey20",
fill = NA, size = 1.2) +
geom_sf(data = Bret_L93, fill = "#440154", colour = "grey60") +
# scale_fill_manual(values = c("#f15522", "#15b7d6", "#ada9ae")) +
coord_sf(crs = 2154, datum = sf::st_crs(2154),
xlim = st_bbox(Bret_buffer30_L93)[c(1,3)],
ylim = st_bbox(Bret_buffer30_L93)[c(2,4)]
) +
theme(title = element_text(size = 16),
panel.background = element_rect(fill = "#66BDFF")) +
ggtitle("Zone tampon autour de la Bretagne")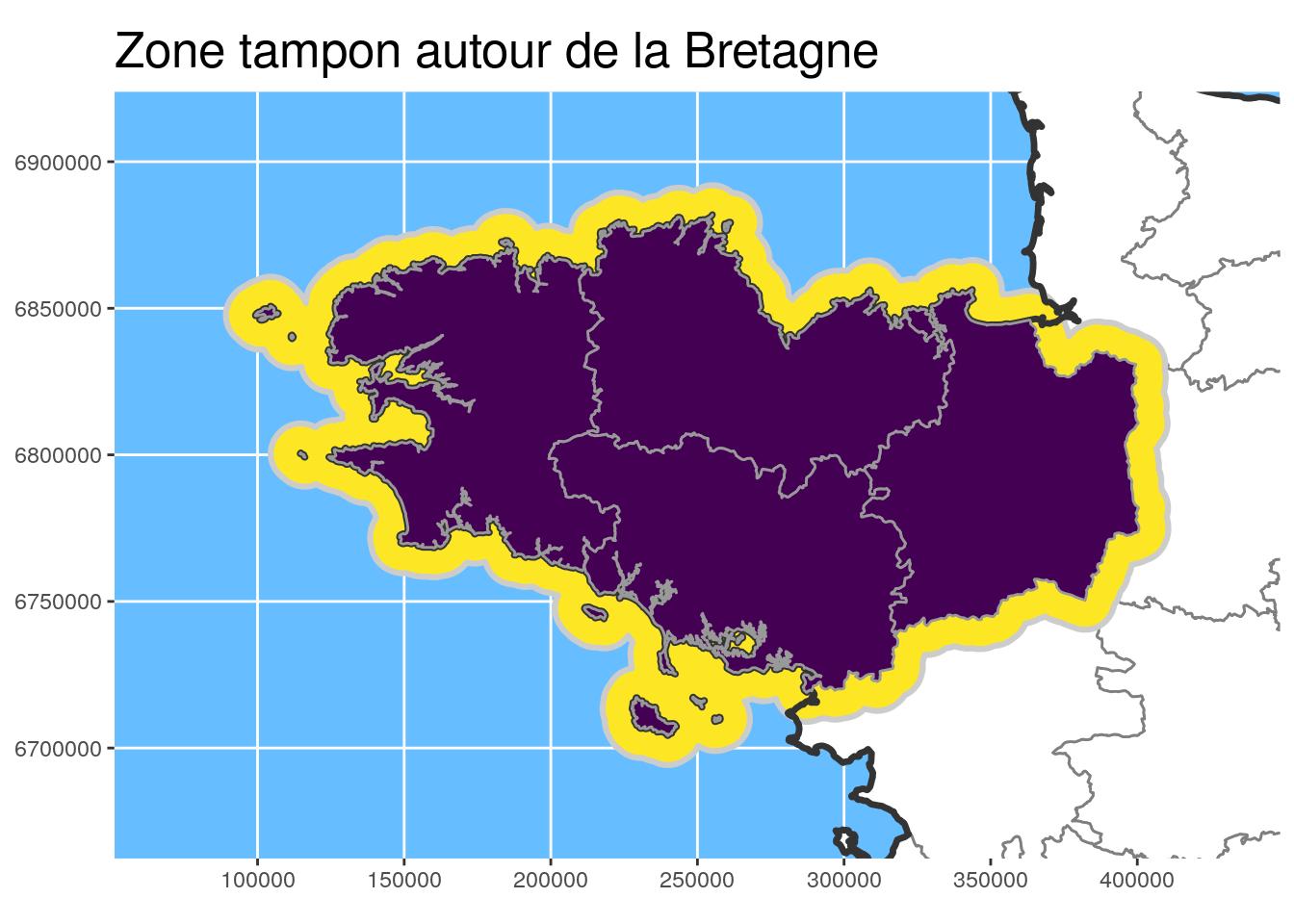
En couplant les zones tampons avec d’autres opérateurs, nous pouvons dessiner des zones de distance à vol de cigogne.
- Récupération des centroïdes de polygones des communes ayant des maternités en Bretagne avec st_centroid
- Unions avec st_union
- Différence avec st_difference
Dans la zone de code ci-dessous, la fonction dist_circles permet de créer ces zones tampons autour d’un point ou de n’importe quel autre objet {sf}.
Fonction
# Define couples distances - years to buffer
#' @param x sf object
#' @param dists c(10000, 25000, 50000, 75000)
#' @param ans unique(maternites_pt$an)
dist_circles <- function(x,
dists = units::set_units(c(10, 25, 50), km),
ans,
x.limits) {
# browser()
if (!missing(ans)) {
dists_ans <- data.frame(
dists = rep(dists, length(ans)),
ans = rep(ans, each = length(dists))
)
# Create buffer areas for each distances / year
pts_buf <- purrr::map2(
dists_ans$dists, dists_ans$ans,
~st_buffer(
filter(x, an == .y),
.x) %>%
mutate(
dist = .x,
)
) %>%
do.call("rbind", .) %>%
st_cast() %>%
mutate(dist.leg = glue::glue("<{dist/1000} km"))
# Define triplet big/small-distance/year
big_small <- data.frame(
big_dist = dists[rep(2:length(dists), length(ans))],
small_dist = dists[rep(1:(length(dists) - 1), length(ans))],
an = ans[rep(1:length(ans), each = length(dists) - 1)]
)
# Remove part of polygons overlapping smaller buffer
pts_holes <- big_small %>%
split(1:nrow(big_small)) %>%
purrr::map(
~st_difference(
filter(pts_buf, dist == .$big_dist, an == .$an),
filter(pts_buf, dist == .$small_dist, an == .$an)
)
) %>%
do.call("rbind", .) %>%
select(-contains(".1")) %>%
st_cast()
# Add smallest polygons and re-order distance names for legend
pts_holes_tot <- pts_holes %>%
rbind(
filter(pts_buf, dist == min(dists))
) %>%
arrange(an, dist) %>%
mutate(dist = forcats::fct_reorder(dist.leg, dist))
} else {
pts_buf <- purrr::map(
dists,
~st_buffer(x, .x) %>%
mutate(dist = .x)
) %>%
do.call("rbind", .) %>%
st_cast() %>%
mutate(dist.leg = as.character(dist))
# Define triplet big/small-distance/year
dists_order_char <- sort(dists) %>% as.character()
big_small <- data.frame(
big_dist = tail(dists_order_char, -1),
small_dist = head(dists_order_char, -1)
)
# Remove part of polygons overlapping smaller buffer
pts_holes <- big_small %>%
split(1:nrow(big_small)) %>%
purrr::map(
~st_difference(
filter(pts_buf, dist.leg == .$big_dist),
filter(pts_buf, dist.leg == .$small_dist) %>%
select(geometry) %>% st_union()
)
) %>%
do.call("rbind", .) %>%
select(-contains(".1")) %>%
st_cast()
# Add smallest polygons and re-order distance names for legend
pts_holes_tot <- pts_holes %>%
rbind(
filter(pts_buf, dist.leg == dists_order_char[1])
) %>%
arrange(dist) %>%
mutate(dist = forcats::fct_reorder(dist.leg, dist))
}
pts_holes_fr <- st_intersection(pts_holes_tot,
dplyr::select(x.limits, geometry))
return(pts_holes_fr)
}Calcul des cercles de distance
# Centroides des communes des maternités
maternites_centroid_Bret_L93 <-
maternites_Bret_L93 %>%
st_centroid()
# Circles around centroids
dists <- c(5, 15, 25, 50)
maternites_circles_L93 <- dist_circles(
maternites_centroid_Bret_L93,
dists = units::set_units(dists, km),
x.limits = Bret_L93)Figure
# Plot and define color according to dist
ggplot() +
geom_sf(data = departements_L93, colour = "grey50",
fill = "white") +
geom_sf(data = st_union(Bret_buffer10_L93), fill = "#FDE725", #"#440154",
colour = "grey80", size = 1.1) +
geom_sf(data = maternites_circles_L93,
aes(fill = dist),
colour = NA) +
scale_fill_brewer(direction = -1) +
geom_sf(data = Bret_L93, fill = NA, colour = "grey60") +
geom_sf(data = st_union(departements_L93), colour = "grey20",
fill = NA, size = 1.2) +
# scale_fill_manual(values = c("#f15522", "#15b7d6", "#ada9ae")) +
coord_sf(crs = 2154, datum = sf::st_crs(2154),
xlim = st_bbox(Bret_buffer30_L93)[c(1,3)],
ylim = st_bbox(Bret_buffer30_L93)[c(2,4)]
) +
theme(title = element_text(size = 16),
panel.background = element_rect(fill = "#66BDFF")) +
ggtitle("Distances à vol d'oiseau du centre\nd'une commune avec maternité")
J’avais utilisé une technique similaire dans l’article “Un halo teinté à l’intérieur d’un polygone avec leaflet et la librairie {sf}”
Dessiner les cartes sur R
Liste personnelle non exhaustive des outils que j’utilise le plus.
Des cartes avec {ggplot2}
Vous avez peut-être remarqué que les cartes précédentes étaient toutes réalisées avec {ggplot2}. C’est maintenant possible avec la dernière version de {ggplot2} (v3.0+) récemment déposée sur le CRAN ! J’ai mis du {viridis} aussi dans les cartes précédentes car c’est aussi possible de manière simple avec scale_fill_viridis_* dans la nouvelle version de {ggplot2}. Si vous voulez en savoir plus sur les nouveautés de la version 3 de {ggplot2}, ThinkR vous en parle !
geom_sf: reconnaît la géométriecoord_sf: limites des axes et CRS- Plusieurs couches peuvent être ajoutées en spécifiant
geom_sf(data = ...)
ggplot() +
geom_sf(data = departements_L93, colour = "grey40",
fill = "white", size = 1.1) +
geom_sf(data = maternites_circles_L93,
aes(fill = dist), colour = NA) +
scale_fill_brewer(direction = -1) +
coord_sf(crs = 2154, datum = sf::st_crs(2154),
xlim = st_bbox(Bret_buffer30_L93)[c(1,3)],
ylim = st_bbox(Bret_buffer30_L93)[c(2,4)]
) +
theme(title = element_text(size = 16),
panel.background = element_rect(fill = "#66BDFF")) +
ggtitle("Distances à vol d'oiseau du centre\nd'une commune avec maternité")
Des cartes avec {tmap}
J’aime bien {tmap} parce que c’est créé pour faire des cartes. Les différents formats de données spatiales sont pris en charge ({sp}, {sf}, {raster}), l’échelle et la flèche du Nord sont gérés nativement. Les couches s’empilent les unes sur les autres à l’aide de tm_shape.
Quelques fonctions :
tm_shapetm_dots: pointstm_fill: polygonestm_text: texte
tm_scale_bartm_compass
# Load background map
# data(Europe) # deprecated in tmap 2.0
data(World)
Europe <- World %>%
filter(continent == "Europe" & name != "France")
# Map
tm <- tm_shape(Europe) +
tm_polygons() +
tm_shape(departements_L93, is.master = TRUE) +
tm_fill(col = "NOM_DEPT", legend.show = FALSE, palette = "Set1") +
tm_borders("grey30") +
tm_shape(maternites_circles_L93) +
tm_fill(col = "dist", palette = "-Blues") +
tm_scale_bar(position = c("right", "bottom"), size = 1) +
tm_compass(position = c("right", "top"), size = 1.8) +
# tm_style_natural() #deprecated in tmap 2.0
tm_style("natural")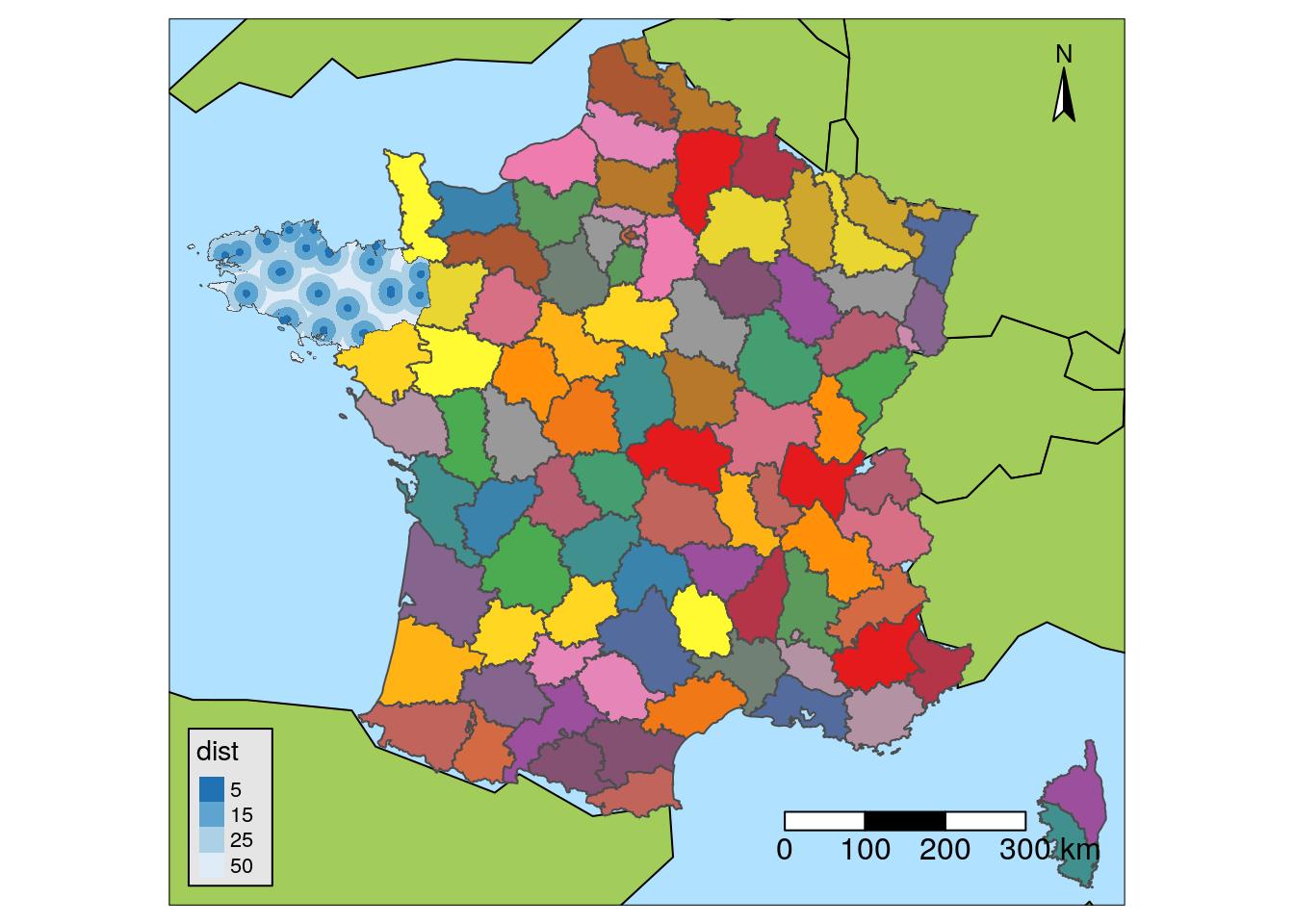
Des cartes interactives avec {leaflet}
Pour proposer des cartes interactives, vous pouvez utiliser {leaflet} :
- Accepte les objets de {sf}
- CRS: 4326
- Créer des palettes pour les couleurs avec
colorFactorpar exemple addTiles(): Fonds de cartesaddMarkers: PointsaddPolygons: Polygones
departements_wgs84 <-
st_transform(departements_L93, crs = 4326)
factpal <- colorFactor(topo.colors(5), departements_wgs84$NOM_REG)
m <- leaflet() %>%
addTiles() %>%
addPolygons(data = departements_wgs84,
color = ~factpal(NOM_REG),
fillOpacity = 0.8, stroke = FALSE)
mDes cartes interactives avec {leaflet} et {tmap}
La magie de {tmap} opère ici : avec tmap_leaflet, vous pouvez transformer votre carte {tmap} en carte interactive ! Pas besoin d’apprendre la syntaxe de leaflet, {tmap} réalise le tour de magie à votre place !
tmap_leaflet(tm)Se passer de QGIS (ou autre) ?
Jusqu’à récemment, il manquait de quoi visualiser et explorer en un coup d’oeil les objets spatiaux produits dans R, de manière interactive. Ce temps est révolu grâce à {mapview} et ses fonctions mapView et viewRGB !
library(mapview)
mapView(departements_L93)Mieux encore, en couplant {mapview} avec {mapedit}, il est maintenant possible de créer et numériser des objets spatiaux directement de manière interactive avec la fonction editMap ! Pourrait-on se passer des logiciels classiques de cartographie tel QGIS ? Le temps le dira… En tout cas, c’est très intéressant pour intégrer de la cartographie interactive dans des Shiny App !
A titre d’exemple, j’ai utilisé ces packages dans l’article de blog pour ThinkR : Le logo ThinkR créé avec la librairie {sf}
logo_pol <- viewRGB(logo) %>% editMap()
logo_overlap <- logo_pol$finished
Et voilà. C’est fini pour cette présentation des outils que j’utilise régulièrement pour faire de la cartographie dans R. Les fonctions proposées par {sf} sont bien plus nombreuses que la dizaine que j’ai présentées ici. Et évidemment, il existe d’autres manières de dessiner des cartes dans R. Vous pourriez regarder les packages {cartography}, {ggmap} ou le récent {ggspatial} par exemple.
L’avenir de la cartographie dans R est adieux !
Now that you know how to create maps with R, you can read these specific articles: Maintenant que vous savez faire de la cartographie avec R, vous pouvez lire ces articles spécifiques :
- Créer des cartes dans R avec {ggplot2}
- Créer des cartes dans R avec {tmap}
- Créer des cartes dans R avec une vue sphérique à l’échelle du globe
Le script R complet de cet article de blog ainsi que les données utilisées se trouvent sur mon dépôt Github “Blog Tips”.
Citation :
Merci de citer ce travail avec :
Rochette Sébastien. (2018, juil.. 14). "Initiation à la cartographie avec {sf} & Co.". Retrieved from https://statnmap.com/fr/2018-07-14-initiation-a-la-cartographie-avec-sf-et-compagnie/.
Citation BibTex :
@misc{Roche2018Initi,
author = {Rochette Sébastien},
title = {Initiation à la cartographie avec {sf} & Co.},
url = {https://statnmap.com/fr/2018-07-14-initiation-a-la-cartographie-avec-sf-et-compagnie/},
year = {2018}
}