Un package R pour la sélection de covariables et la modélisation des distributions d’espèces
J’ai décidé de transformer en un package R, des scripts que j’utilise depuis des années pour mes recherches avec la modélisation de distribution d’espèces. Je sais que ce package peut être amélioré de multiples manières mais cette v0.1 fonctionne en l’état.
Soyez sympa, c’est mon premier vrai package R ! Souvenez-vous que c’est une compilation rapide de différents codes qui ont évolués au cours de ces dernières années (lesquelles avaient des standards de codage différents…). Cela dit, je suis sûr que la communauté R trouvera de nombreuses manières (et du temps ?) pour l’améliorer.
Cette librairie a été initialement conçue pour la sélection de covariables en vue d’ajuster des modèles d’habitat (biomasses, densités ou présence/absence). Son objectif premier est donc de produire des cartes de prédiction de distributions (cf. vignette SDM_Selection). Cependant, le coeur de la librarie est une validation croisée “k-parties N-fois” qui peut s’appliquer à n’importe quel type de données, si les paramètres de la procédure sont correctement définis (cf. vignette Covar_Selection).
La librairie fonctionne avec les dataframe, SpatialPointsDataFrame et tibbles.
Elle a été conçue pour fonctionner en parallèle sur un ordinateur multicoeur, grâce à la librairie parallel.
Les seules étapes manquantes de la librairie SDMSelect sont l’exploration et le nettoyage des données. Ce sont des parties importante de la modélisation que vous devriez réaliser avant la procédure de sélection de modèle.
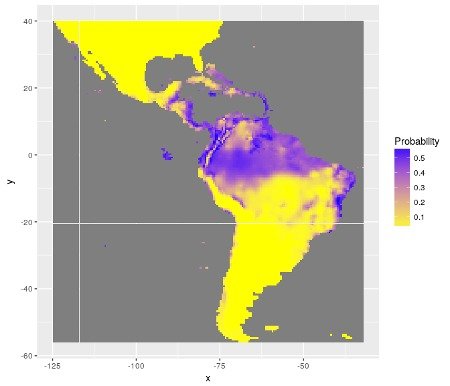
Figure 1: Distribution de probabilités de présence
Méthode de sélection de covariables
La procédure de sélection de modèles testera les différentes combinaisons de covariables dans des modèles LM, GLM, GLM avec splines naturelles et GAM, avec différentes distributions (Gaussienne, Gamma, Log-Normal, Tweedie; Binomiale) et avec différents degrés de liberté maximum pour les GLM avec polynômes ou les splines naturelles.
La validation croisée en k-parties répétée N-fois sur les mêmes sous-parties du jeu de données pour chaque type de modèle ou sous-modèle permet de comparer tous ces types entre eux par RMSE ou AUC. La corrélation entre les covariables peut être testée avant la procédure de sélection pour éviter l’ajustement de modèles intégrant des covariables corrélées. Cependant, la procédure de validation croisée et couplée avec une procédure d’ajout de covariables progressive. Cela signifie que les covariables sont ajoutées au meilleur modèle sélectionné à l’étape précédente. Cette procédure ne retient pas l’ajout de covariables qui n’apportent pas de pouvoir prédictif au modèle, et doit donc empêcher la sélection de covariables corrélées dans un même modèle.
Les modèles sont ordonnés en fonction du RMSE (ou de l’AUC pour la présence-absence). La procédure de validation croisée produit une distribution de (N*k) valeurs de RMSE pour chaque modèle estimé à chaque étape. Les modèles sont rangés en fonction du RMSE moyen. Les distributions de RMSE des modèles n°2 à n sont ensuite comparées statistiquement (wilcoxon) à celle du meilleur modèle. Les modèles qui ne sont pas statistiquement plus mauvais que le meilleur modèle sont retenus pour l’étape suivante. À la fin de la procédure pas-à-pas progressive, le meilleur modèle d’entre tous est retenu par la même méthode. Les modèles n’étant pas statistiquement plus mauvais que le meilleur sont aussi identifiés.
Sorties
Les sorties de la procédure de sélection sont nombreux. Ils permettent de résumer la sélection de modèles et de comparer la totalité des différents modèles ajustés entre eux (GAM, GLM; Gaussian, Gamma, Log-Normal, Tweedie; Binomial). Tous les fichiers et figures de sortie sont enregistrés dans un dossier commun et n’apparaissent pas directement dans la session R. Le modèle finalement retenu par l’utilisateur peut ensuite être analysé (analyse de résidus, analyse de variance, effet des covariables). Pour les données spatialisées, des cartes de prédiction sont aussi produites.
Incertitudes
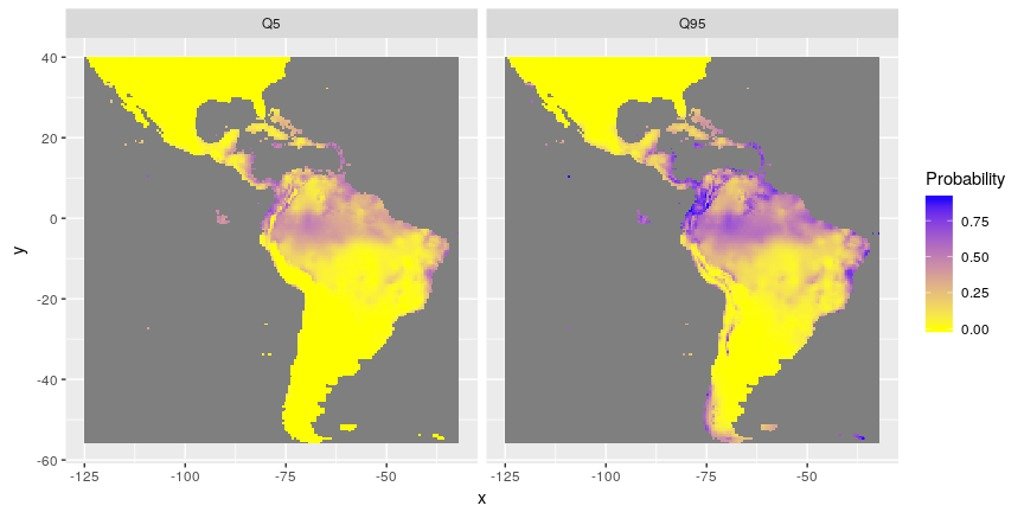
Une attention particulière a été portée à l’évaluation de l’incertitude. En effet, chaque prédiction d’un modèle est donnée avec une erreur standard associée. Ces erreurs standard ont été utilisées pour estimer les distributions d’espèces minimum et maximum (au travers de l’estimation des quantiles).
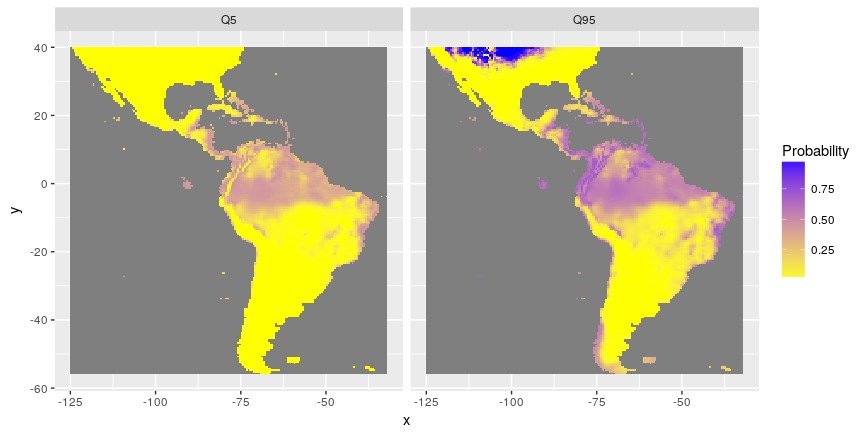
Figure 2: Distribution des minimum (quantile 5%) et maximum (quantile 95%) de probabilités de présence
Concernant les données de présence-absence, l’estimation de la probabilité de présence n’est pas suffisante. La balance entre les présences et les absences dans le jeu de données peut conduire à l’estimation de prédictions biaisées. Le meilleur seuil permettant de classer les probabilités de présence en présence ou absence est ici exploré plus en détail. Une carte de probabilité d’être au-dessus du meilleur seuil est calculée.
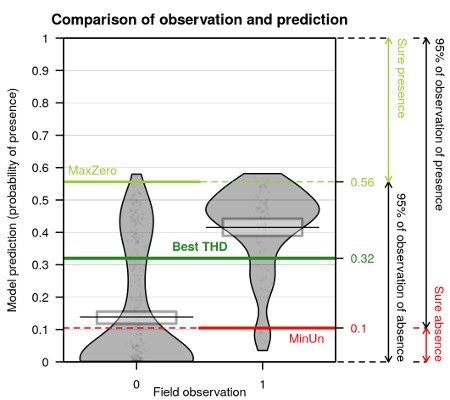
Figure 3: Comparaison des prédictions avec les observations dans un modèle de présence-absence et affichage des différents seuils
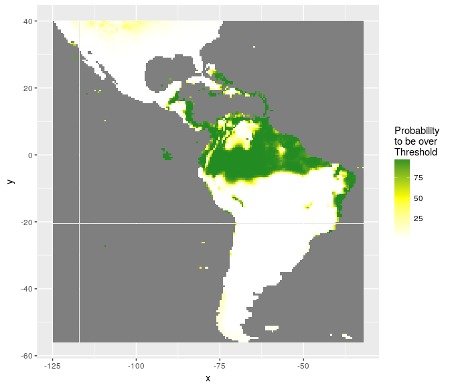
Figure 4: Distribution des probabilités d’être au-dessus du meilleur seuil séparant les présences des absences
Précautions
Cette librairie repose sur un grand nombre d’autres librairies R, ce qui signifie que chaque modification de ces librairies peut empêcher SDMSelect de fonctionner correctement. Comme j’utilise cette librairie régulièrement, je devrais normalement être amené à corriger les éventuels problèmes.
Il existe maintenant de nouvelles librairies clés dans R comme dplyr, tidyr, ggplot2 qui n’existaient pas lorsque j’ai créé ces scripts. Je les intègre à l’occasion.
Remarques
Cette librairie n’est pas aussi complète que peut l’être la librairie caret en terme de types de modèles disponibles, cependant, il me semble que la procédure de sélection de la librairie caret n’a été définie que pour une unique combinaison modèle/distribution. Ici, les sorties de validation croisée de chaque combinaison modèle/distribution sont retenues et peuvent être comparées toutes ensemble de manière pairée. Par ailleurs, non seulement le modèle avec le meilleur RMSE moyen (ou AUC) est retenu, mais aussi tous les autres modèles produisant des prédictions statistiquement aussi bonnes que le meilleur modèle. Dans le cadre d’une étude en biologie, connaître tous les modèles ayant un pouvoir prédictif équivalent peut modifier l’interprétation des résultats. Une autre différence est que la librairie SDMSelect a été construite pour la prédiction de distribution d’espèces et produit donc des cartes de distributions ainsi que toutes les cartes d’incertitude de prédiction qui peuvent être déduites des sorties de modèles.
Collaboration pour la modélisation de distribution d’espèces
Pour vos travaux de sélection de covariables et la modélisation des distributions d’espèces, vous pouvez me contacter. Les vignettes du package sont un bon point de départ à l’utilisation de la librairie. Je participerai cependant volontiers à une collaboration scientifique impliquant l’utilisation de cette librairie.
Si vous souhaitez participer à l’amélioration de cette librairie, vous pouvez jeter un oeil à ma “todo list” sur github. N’hésitez pas à cloner, modifier et envoyer des requêtes de modification sur le github de SDMSelect.
Télécharger et installer
Avant d’installer SDMSelect, vérifiez que vous avez les versions mises à jour de mgcv (>= 1.8-19) et dplyr (>= 0.7). De plus, corrplot doit être installé depuis son dépôt github pour avoir la version taiyun/corrplot (>= 0.82): devtools::install_github("taiyun/corrplot").
Pour télécharger la version de développement de la librairie SDMSelect utiliser la commande suivante:
install.packages("devtools")
devtools::install_github("statnmap/SDMSelect")Pour pouvoir voir les vignettes, vous devrez installer knitr, rmarkdown, dismo et rasterVis, et installer SDMSelect avec:
devtools::install_github("statnmap/SDMSelect", build_vignettes = TRUE, dependencies = c("Depends", "Imports", "Suggests"))Notez que les librairies telles que rgdal et sp peuvent nécessiter des logiciels supplémentaires que vous devrez installer sur votre ordinateur si vous travaillez sur Mac ou Linux. Regardez comment installer proj4, geos et gdal sur votre système.
Exemples
Je pense écrire quelques autres posts de blog pour mieux expliquer les différentes fonctions de cette librairie mais les deux vignettes incluses vous montrerons les principales. Les vignettes ont été créées pour montrer comment utiliser la librairie sur des cas simples de sélection de covariables (vignette(package = "SDMSelect")).
- Le premier cas est une procédure de sélection pour un jeu de données classique (pas de données géographiques, pas de distribution d’espèces):
vignette("Covar_Selection", package = "SDMSelect"). Vous pouvez la lire dans la page {pkgdown} “covar selection”
- Le second cas utilise des données géographiques pour produire une carte de prédiction de distribution d’espèce et les cartes d’incertitude associées:
vignette("SDM_Selection", package = "SDMSelect"). Vous pouvez la lire dans la page {pkgdown} “SDM selection”
Notez que la plupart des figures des vignettes sont sauvées dans le dossier “inst” de la librairie, de telle sorte que la sélection de modèles ne soit pas effectuée au moment de la construction de la vignette. Cependant, le code écrit dans les vignettes peut être entièrement lancé sur votre ordinateur et devrait sortir les mêmes résultats. Vous pouvez aussi trouver les fichiers complets des vignettes à faire tourner en un clic sur votre ordinateur: system.file("Covar_Selection", "Covar_Selection.Rmd", package = "SDMSelect") et system.file("SDM_Selection", "SDM_Selection.Rmd", package = "SDMSelect"). Ouvrir et cliquer sur le bouton knit si vous êtes sur Rstudio (Cela devrait prendre 5-10 minutes selon le nombre de processeurs de votre ordinateur).
La plupart des fonctions sont listées dans l’aide générale de la librairie : ?SDMSelect.
Licence
Ce package est distribué avec une licence libre et open-source, sous GPL.
Remerciements
Ce travail a été en partie permi par mon implication dans ATLAS, un programme Recherche et Innovation (Horizon 2020) de l’Union Européenne ayant l’accord de subvention No 678760. Les résultats reflètent uniquement l’opinion de l’auteur et l’Union Européenne ne peut en aucun cas être tenue responsable de toute utilisation qui pourrait être faite de l’information qu’ils contiennent.
Citation :
Merci de citer ce travail avec :
Rochette Sébastien. (2017, sept.. 23). "SDMSelect: Sélection de modèle par validation croisée et cartographie des distributions d'espèces". Retrieved from https://statnmap.com/fr/2017-09-23-sdmselect-package-modelisation-distribution-d-especes/.
Citation BibTex :
@misc{Roche2017SDMSe,
author = {Rochette Sébastien},
title = {SDMSelect: Sélection de modèle par validation croisée et cartographie des distributions d'espèces},
url = {https://statnmap.com/fr/2017-09-23-sdmselect-package-modelisation-distribution-d-especes/},
year = {2017}
}

Comment citer
Merci de remercier et citer les packages R lorsque vous les utilisez pour vos analyses de données. Pour savoir comment citer
SDMSelectcorrectement, utilisez la commandecitation("SDMSelect"):