Une simple boucle pour ajouter plusieurs couches
La librarie leaflet s’intègre très bien avec l’écriture façon “pipe” (%>%). Il est donc très facile d’ajouter de multiple couches spatiales dans un widget leaflet.
Pour ajouter de nombreuses couches spatiales à votre widget, il n’est pas nécessaire d’utiliser une fonction comme “addPolygons” pour chacune. Vous pouvez simplement utiliser une boucle. Vous aurez d’abord besoin de standardiser les noms de vos fichiers de couches pour avoir un code clair. La standardisation vous permet aussi de créer différents groupes avec des options d’affichage différentes.
Le widget leaflet suivant est un example d’affichage de multiples couches spatiales qui ont été ajoutées avec une boucle. Le code R est disponible sur mon github.
Fixation des quotas de pêche 2017
Pour de trop nombreux stocks, les ministres ne pourront pas suivre l’avis scientifique
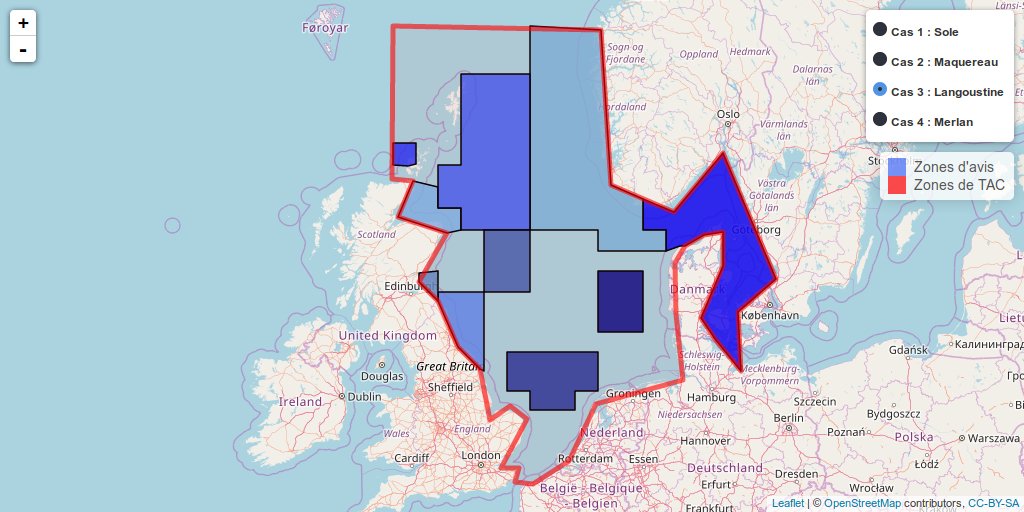
L’exemple ci-dessous est un widget réalisé pour l’AFH (Association Française d’Halieutique). Il montre différent cas de discordance entre les zones de quotas définies par la commission européenne et les zones délimitées par les scientifiques pour donner leur avis sur l’état des ressources. La note complète peut être téléchargée directement sur le site Internet de l’AFH.
Points clés de la note de l’AFH
- Les 12 et 13 décembre 2016, les ministres des pêches de l’UE fixeront les quotas 2017, en principe en conformité avec les avis des scientifiques et avec l’objectif d’une pêche durable, capable de fournir le rendement maximum équilibré.
- Les avis scientifiques sont établis à l’échelle de stocks qui sont des unités biologiques fonctionnelles, dont les contours évoluent au fur et à mesure de l’avancée des connaissances.
- Les quotas de pêche se réfèrent quant à eux à des unités de gestion, fixées en 1982 et qui n’ont pas ou peu évolué depuis.
- Il existe ainsi une discordance croissante entre la réalité biologique et les mesures de gestion mises en œuvre. Il s’ensuit un processus de fixation des quotas très complexe et souvent opaque pour l’ensemble des acteurs.
- Et surtout, ces discordances sont source de risques pour la durabilité de la ressource biologique. Elles compromettent, pour les stocks concernés, l’objectif d’une gestion au rendement maximum durable.
Widget leaflet
- Cas 1: concordance unité de gestion – unité fonctionnelle
- Cas 2 : L’avis scientifique donne lieu à plusieurs Totaux Admissibles de Capture (TAC)
- Cas 3 : Un TAC regroupe plusieurs avis
- Cas 4 : Déconnexion entre unité de gestion et unité d’avis scientifique (découpage et/ou recombinaison de zones d’avis, mélange des cas précédents)
R-code to loop multiple layers in a leaflet widget
library(leaflet)
library(rgdal)
# Working directory ----
wd <- ""
# Read multiple shapefiles with standardised name ----
species <- c("Sole", "Maquerel", "Langoustine", "Merlan")
Groupnames <- c("Cas 1 : Sole", "Cas 2 : Maquereau", "Cas 3 : Langoustine",
"Cas 4 : Merlan")
for (sp in species) {
for (type in c("Avis", "TAC")) {
files.sp <- readOGR(dsn = wd, layer = paste(sp, type, sep = "_"),
verbose = FALSE)
assign(paste(sp, type, sep = "_"), files.sp)
}
}
# Create leaflet widget --------------------------------------------------------
m <- leaflet() %>%
addTiles(group = "OSM (default)")
# Add multiple layers with a loop ----------------------------------------------
for (sp.N in 1:length(species)) {
sp <- species[sp.N]
for (type in c("Avis", "TAC")) {
tmp <- get(paste(sp, type, sep = "_"))
# Define different colors depending on type of data
if (type == "Avis") {
myPal <- colorRampPalette(c("blue", "skyblue", "navyblue"))
factpal.Div <- colorFactor(myPal(length(tmp$id)), tmp$id)
m <- m %>%
addPolygons(data = tmp,
fillColor = ~factpal.Div(tmp$id),
color = "#000000",
opacity = 1,
fillOpacity = 0.8,
stroke = TRUE,
weight = 1.5,
smoothFactor = 0.2,
popup = paste0("Zone d'avis : ", tmp$id),
group = Groupnames[sp.N]
)
} else {
myPal <- colorRampPalette(c("red", "orange", "brown"))
factpal.Div <- colorFactor(myPal(length(tmp$id)), tmp$id)
m <- m %>%
addPolygons(data = tmp,
fillColor = "#FF0000",
color = ~factpal.Div(tmp$id),
opacity = 0.6,
fillOpacity = 0.05,
stroke = TRUE,
weight = 5,
smoothFactor = 0.2,
popup = paste0("Zone de TAC : ", tmp$id),
group = Groupnames[sp.N]
)
}
}
} # end of species
# Additional leaflet options ---------------------------------------------------
m <- m %>%
# Add layers controls
addLayersControl(
baseGroups = Groupnames,
options = layersControlOptions(collapsed = FALSE)
) %>%
# Add common legend
addLegend(colors = c("#4367F4", "#FF0000"),
labels = c("Zones d'avis", "Zones de TAC"),
opacity = c(0.8, 0.5))
# Print the map ----------------------------------------------------------------
mCitation :
Merci de citer ce travail avec :
Rochette Sébastien. (2016, déc.. 01). "Inclure plusieurs couches dans un widget leaflet avec R". Retrieved from https://statnmap.com/fr/2016-12-01-plusieurs-couches-dans-widget-leaflet-avec-rstat/.
Citation BibTex :
@misc{Roche2016Inclu,
author = {Rochette Sébastien},
title = {Inclure plusieurs couches dans un widget leaflet avec R},
url = {https://statnmap.com/fr/2016-12-01-plusieurs-couches-dans-widget-leaflet-avec-rstat/},
year = {2016}
}