Corrélation entre objets raster
Dans cet article de blog, je propose une façon d’aborder la répartition spatiale de la corrélation entre deux rasters. Quelque soit votre raison, vous pourriez vouloir connaître la corrélation entre des rasters, sauf qu’une valeur unique issue de la fonction cor n’est pas suffisante. Vous aimeriez savoir s’il existe des variations de corrélation dans l’espace. Vous ne pouvez pas comparer les raster point par point, alors utilisons une petite astuce.
Quelques utilitaires pour les figures
Pour dessiner un raster avec {ggplot2}, j’ai récemment modifié la fonction gplot de la bibliothèque {rasterVis} afin que nous puissions récupérer les valeurs raster extraites sous forme de tibble, au lieu de faire la figure directement. Entre autres, cela permet d’enlever des cellules sans valeurs (valeur = NA), ce qui rend le raster plus léger à tracer. Cette fonction est appelée gplot_data dans mon {[paquet SDMSelect sur github](//github. com/statnmap/SDMSelect}. Je le présente un peu dans cette section du blog.
Par ailleurs, nous ajouterons des polygones de pays européens. J’ai trouvé une partie de la liste sur le site d’Ewen Gallic, ce qui m’a sauvé car je ne suis pas sûr que j’aurai eu la liste complète…
# Libraries
library(raster)
library(dplyr)
library(mapview)
library(mapedit)
library(sf)
library(readr)
library(ggplot2)Comparaison entre la Température de l’eau et la Chlorophylle-a
Pour l’exemple, nous utilisons des rasters (1) de la température moyenne du fond au printemps de 2010-2014, extraites du modèle hydrodynamique MARS3D (Ifremer, Cailleaux et al., 2016. Données du modèle 500m.) et (2) de la concentration moyenne de chlorophylle en surface au printemps de 2003-2010, extraite du satellite MODIS (Ifremer, Gohin, F., Druon, J. N., and Lampert, L.: A five channel chlorophyll concentration algorithm applied to SeaWiFS data processed by Seadas in coastal waters, (2002). International Journal of Remote Sensing, 23, 1639-1661.
Désolé, ces données ne sont pas directement disponibles pour faire un exemple reproductible, mais si vous souhaitez ces données là en particulier, vous pouvez demander aux auteurs ci-dessus.
temp_r <- raster(file.path(extraWD, "PREVIMER_F1-MARS3D-MANGAE2500_2010-2014_spring_TEMP_mean.tif"))
chl_r <- raster(file.path(extraWD, "chlorophyll_a_2003-2010_spring_mean.tif"))
# Geographic Western Europe
Europe <- c(
"Austria", "Belgium", "Bulgaria", "Croatia", "Cyprus",
"Czech Rep.", "Denmark", "Estonia", "Finland", "France",
"Germany", "Greece", "Hungary", "Ireland", "Italy", "Latvia",
"Lithuania", "Luxembourg", "Malta", "Netherlands", "Poland",
"Portugal", "Romania", "Slovakia", "Slovenia", "Spain",
"Sweden", "UK",
"Switzerland", "Norway", "Monaco", "Jersey", "Guernsey",
"Azores")
Europe_border <- map_data("world") %>%
filter(region %in% Europe)
# This extract part of the data for lighter plots (like rasterVis)
temp_dat <- SDMSelect::gplot_data(temp_r, maxpixels = 50000) %>%
mutate(variable = "Temperature") %>%
filter(!is.na(value))
chl_r_dat <- SDMSelect::gplot_data(chl_r, maxpixels = 50000) %>%
mutate(variable = "Chlorophyll-a") %>%
filter(!is.na(value))
# Plot
g1 <- ggplot(temp_dat) +
geom_tile(aes(x, y, fill = value)) +
scale_fill_gradient2("T°C",
low = scales::muted("blue"),
high = scales::muted("red"),
midpoint = mean(temp_dat$value)) +
geom_polygon(data = Europe_border,
aes(long, lat, group = group),
fill = "white",
colour = "grey20", size = 0.1) +
coord_quickmap(
xlim = range(temp_dat$x),
ylim = range(temp_dat$y)
) + xlab("") + ylab("")
g2 <- ggplot(chl_r_dat) +
geom_tile(aes(x, y, fill = value)) +
scale_fill_gradient("Chl-a", low = "white",
high = "forestgreen",
trans = "log") +
geom_polygon(data = Europe_border,
aes(long, lat, group = group),
fill = "white",
colour = "grey20", size = 0.1) +
coord_quickmap(
xlim = range(chl_r_dat$x),
ylim = range(chl_r_dat$y)
) + xlab("") + ylab("")
gridExtra::grid.arrange(g1, g2, ncol = 2)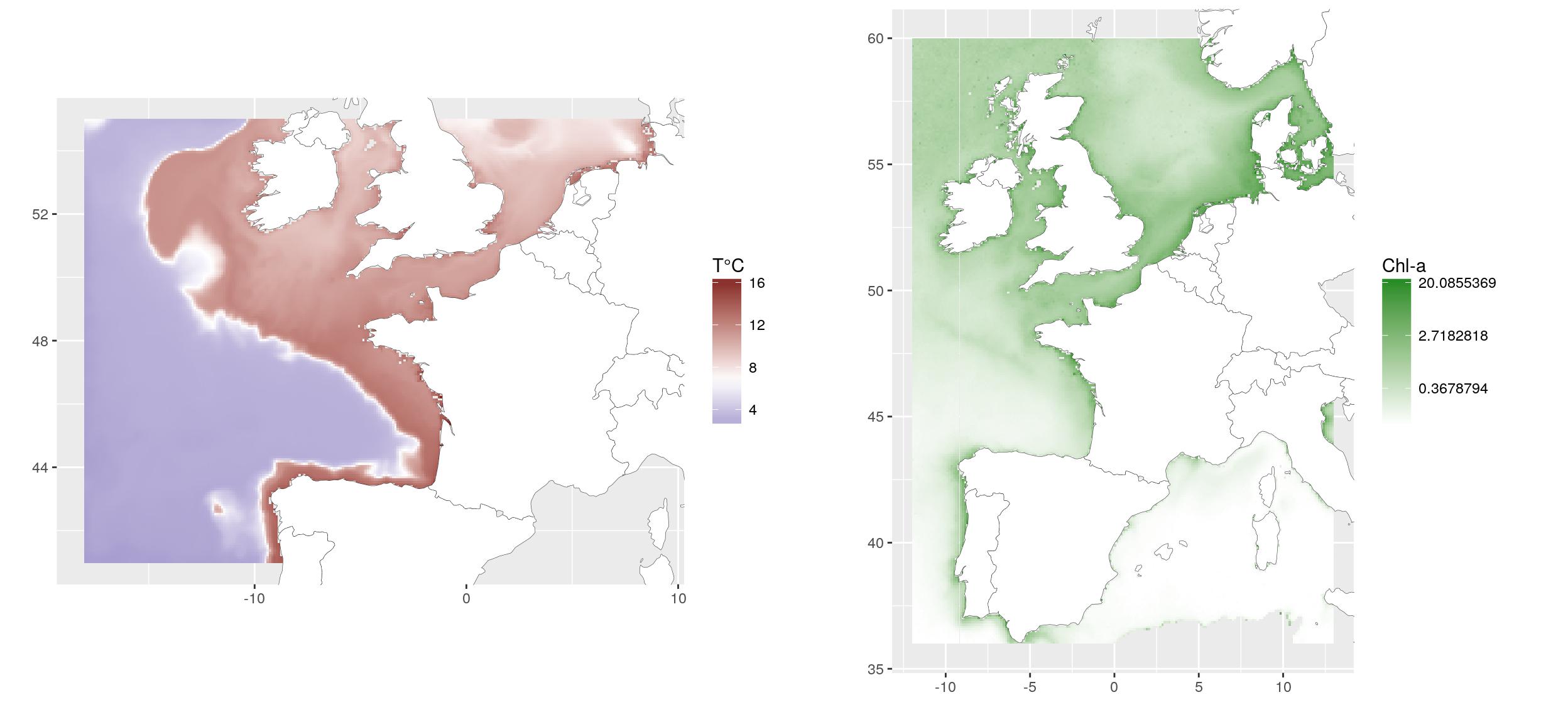
Agréger, recadrer et rééchantillonner les rasters pour permettre la comparaison
Parce que j’aime {mapview} et {mapedit}, je les utilise pour définir une zone d’étude réduite sur laquelle travailler. J’ai déjà joué avec ces bibliothèques spatiales sur un billet de blog pour ThinkR.
ext_pol <- mapview(temp_r) %>%
editMap()
extent_pol <- ext_pol$finishedwrite_rds(extent_pol, file.path(extraWD, "extent_pol.rds"))Nous pouvons maintenant recadrer le raster de température en utilisant l’étendue définie. Je l’ai aussi agrégé (x2) pour avoir moins de données et accélérer les calculs qui vont suivre. Comme le polygone d’étendue a été créé comme un objet {sf}, nous devons le convertir en objet {sp} pour le rendre compatible avec les fonctions de bibliothèque {raster}. Le raster de chlorophylle est également agrégé. J’ai utilisé une agrégation x4 pour que sa résolution soit plus proche de celle de la température. Cela peut être important compte-tenu de la façon dont la fonction resample fonctionne, mais je n’entrerai pas dans les détails. Ensuite, je peux rééchantillonner le raster de chlorophylle pour qu’il soit totalement comparable à celui de la température.
temp_crop_r <- raster::crop(temp_r, as_Spatial(st_geometry(extent_pol))) %>%
aggregate(2)
chl_res_r <- aggregate(chl_r, 4) %>%
resample(temp_crop_r)
# Stack covariates
temp_chl_s <- stack(temp_crop_r, chl_res_r)
names(temp_chl_s) <- c("temperature", "chlorophyll-a")
# Old way to plot...
plot(temp_chl_s)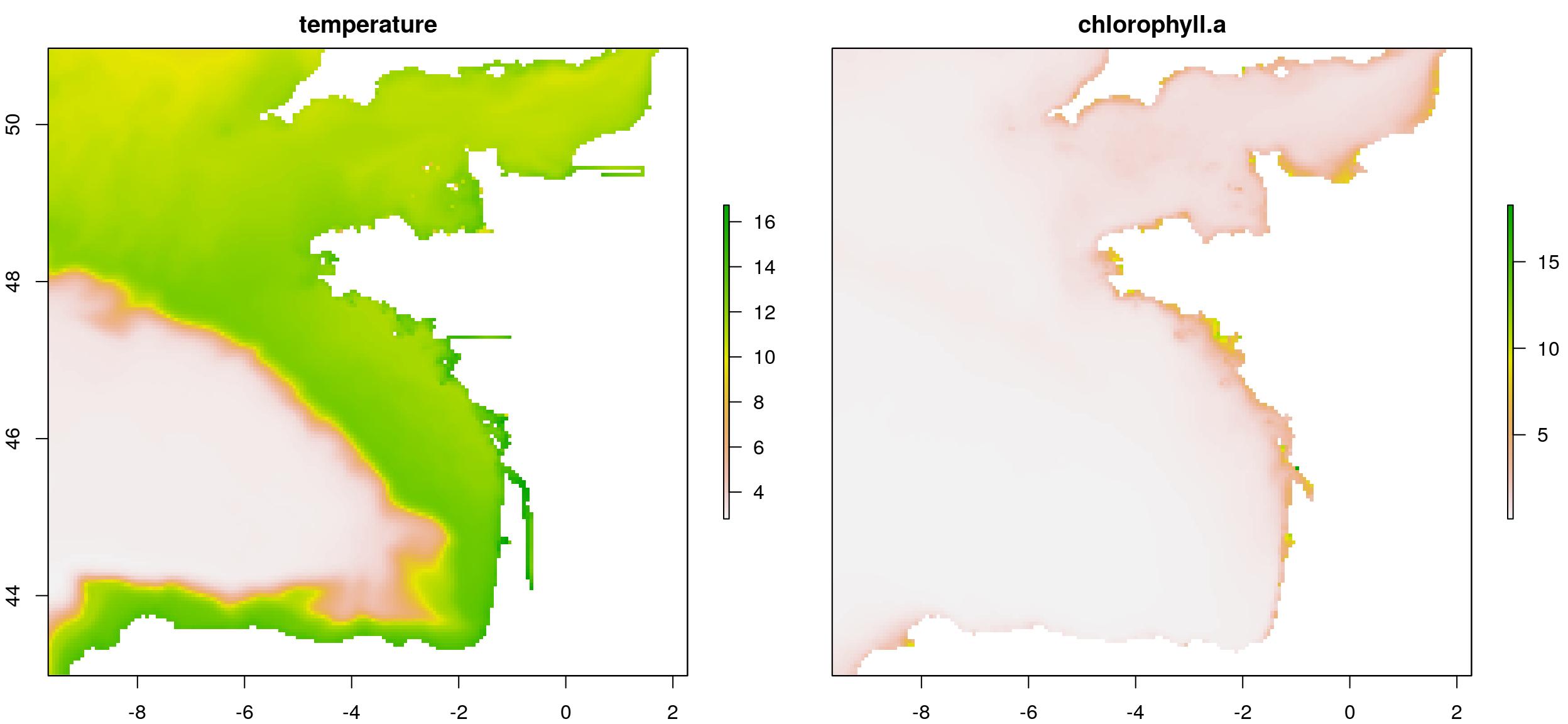
Calculer la corrélation globale
La corrélation globale est une opération habituelle utilisant la fonction cor entre les valeurs des deux rasters. Elle ne renvoie qu’une seule valeur.
# Correlation between layers
cor(values(temp_chl_s)[,1],
values(temp_chl_s)[,2],
use = "na.or.complete")## [1] 0.3945855Estimer un modèle linéaire
Une autre façon de voir s’il y a une corrélation entre les deux couches est de définir un modèle linéaire (ici lm (chl-a ~ T°C)). Pour examiner l’hétérogénéité spatiale, nous pouvons tracer la distribution des résidus du modèle. (Ne pas oublier de récupérer les résidus manquants des valeurs NA.). Cette carte montre où les observations s’écartent du modèle. On pourrait dire que la Chlorophylle-a est sous-estimée le long des côtes lorsqu’on utilise la température comme prédicteur linéaire.
lm1 <- lm(values(temp_chl_s)[,2] ~ values(temp_chl_s)[,1])
summary(lm1)##
## Call:
## lm(formula = values(temp_chl_s)[, 2] ~ values(temp_chl_s)[, 1])
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.7938 -0.4277 -0.0068 0.0679 10.6740
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.128173 0.013327 -9.617 <2e-16 ***
## values(temp_chl_s)[, 1] 0.084055 0.001383 60.794 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.7664 on 20042 degrees of freedom
## (11462 observations deleted due to missingness)
## Multiple R-squared: 0.1557, Adjusted R-squared: 0.1557
## F-statistic: 3696 on 1 and 20042 DF, p-value: < 2.2e-16# Retrieve residuals considering missing values
resid_lm <- raster(temp_chl_s, 1) * NA
values(resid_lm)[-lm1$na.action] <- lm1$residuals
# Figure
resid_lm_dat <- SDMSelect::gplot_data(
resid_lm, maxpixels = 50000) %>%
mutate(variable = "Residuals") %>%
filter(!is.na(value))
ggplot(resid_lm_dat) +
geom_tile(aes(x, y, fill = value)) +
scale_fill_gradient2("Residuals",
low = scales::muted("red"),
high = scales::muted("blue"),
midpoint = 0) +
geom_polygon(data = Europe_border,
aes(long, lat, group = group),
fill = "white",
colour = "grey20", size = 0.1) +
coord_quickmap(
xlim = range(resid_lm_dat$x),
ylim = range(resid_lm_dat$y)
) + xlab("") + ylab("")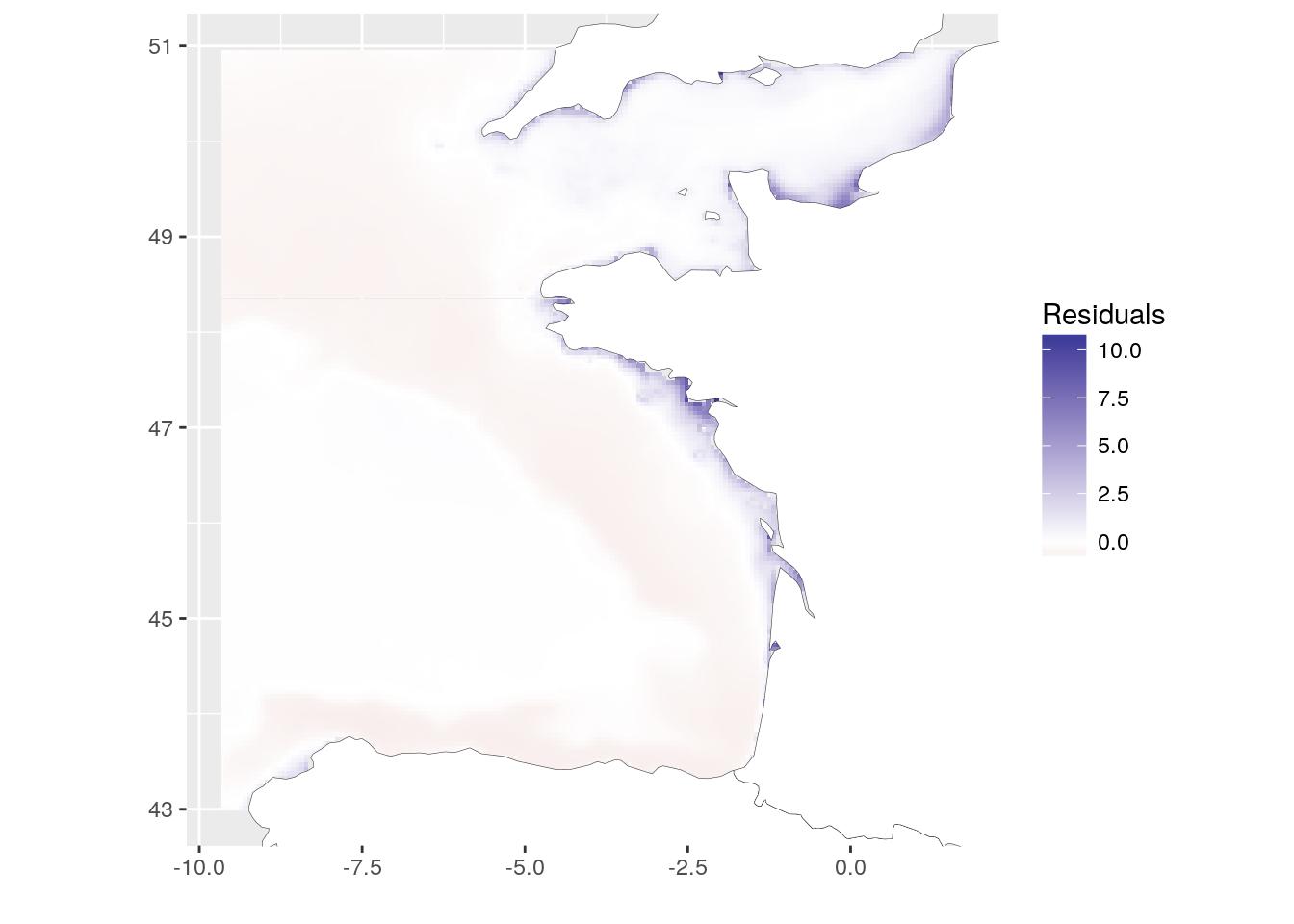
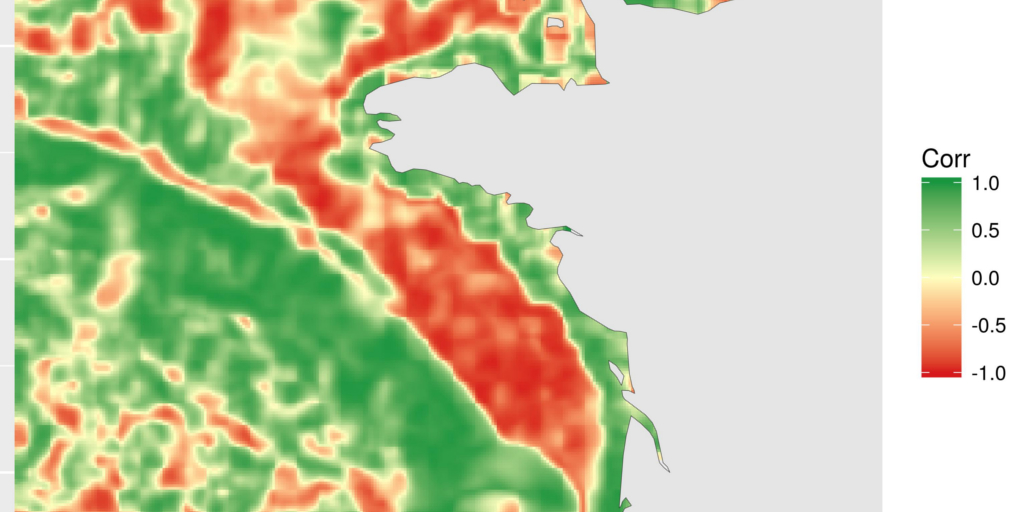
Calculer la corrélation locale en utilisant focal sur deux rasters en même temps
La corrélation locale est une façon différente de voir la relation entre deux covariables dans l’espace. L’idée est simple. Autour de chaque cellule des rasters, nous définissons une zone focale (carrée, ronde,…). Pour l’exemple, j’ai choisi un carré de 5*5. Nous calculons la corrélation entre les 25 valeurs de chaque raster issues de ce carré en utilisant la fonction cor. Cette corrélation locale est enregistrée pour la cellule centrale. Cette opération est répétée sur la totalité du raster grâce à la fonction focal de la librairie {raster}.
Cependant, nous devons utiliser une astuce pour utiliser la fonction sur deux rasters en même temps. Nous utilisons un troisième raster (temp_chl_s_nb) qui enregistre les positions des valeurs de 1 à ncell. La fonction focal va donc extraire les positions pour lesquelles on peut obtenir les valeurs des deux rasters (puisqu’ils ont maintenant les mêmes étendue, origine et résolution).
[EDIT 2019-09-07] Grâce à un commentaire de Matthias Weigand, on peut accelérer la procédure en passant le stack en matrice dans la fonction focal. Voir les lignes avec le commentaire [MW] ci-dessous. Soyez prudents toutefois si vous manipulez de gros rasters, cela pourrait demander plus de RAM.
temp_chl_s_nb <- raster(temp_chl_s, 1)
values(temp_chl_s_nb) <- 1:ncell(temp_chl_s)
matrix_chl_s <- values(temp_chl_s) # stack as raster [MW]
focal_cor <- focal(
x = temp_chl_s_nb,
w = matrix(1, 5, 5),
# fun = function(x, y = temp_chl_s){ # Original
# cor(values(y)[x, 1], values(y)[x, 2], # Original
# use = "na.or.complete")
# },
fun = function(x, y = matrix_chl_s){ # [MW]
cor(y[x, 1], y[x, 2], # [MW]
use = "na.or.complete")
},
filename = file.path(extraWD, "focal_cor.tif"),
overwrite = TRUE
)La carte produite montre les zones où la corrélation entre la chlorophylle-a et la température est négative, nulle ou positive. C’est une façon différente de voir la relation entre les covariables. Ici, par exemple, on peut voir la relation positive le long des côtes et la relation négative sur le plateau. Je n’entrerai pas ici dans des considérations scientifiques plus poussées. Ce n’est pas le but de cet article. Je vous rappelle aussi que les covariables sont la température de fond, telle que modélisée par un modèle hydrodynamique 3D et la chlorophylle de surface, telle que calculée à partir des données satellitaires. La comparaison générale n’a probablement qu’un faible intérêt scientifique, mais ce sont les covariables (ayant une étendue différente) les plus accessibles que j’avais avec moi aujourd’hui…
focal_cor <- raster(file.path(extraWD, "focal_cor.tif"))
# Get data for ggplot
focal_cor_dat <- SDMSelect::gplot_data(focal_cor, maxpixels = 50000) %>%
mutate(variable = "Correlation") %>%
filter(!is.na(value))
# Plot
ggplot(focal_cor_dat) +
geom_tile(aes(x, y, fill = value)) +
scale_fill_gradient2("Corr",
low = "#d7191c",
mid = "#ffffbf",
high = "#1a9641",
midpoint = 0) +
geom_polygon(data = Europe_border,
aes(long, lat, group = group),
fill = "white",
colour = "grey20", size = 0.1) +
coord_quickmap(
xlim = range(focal_cor_dat$x),
ylim = range(focal_cor_dat$y)
) + xlab("") + ylab("")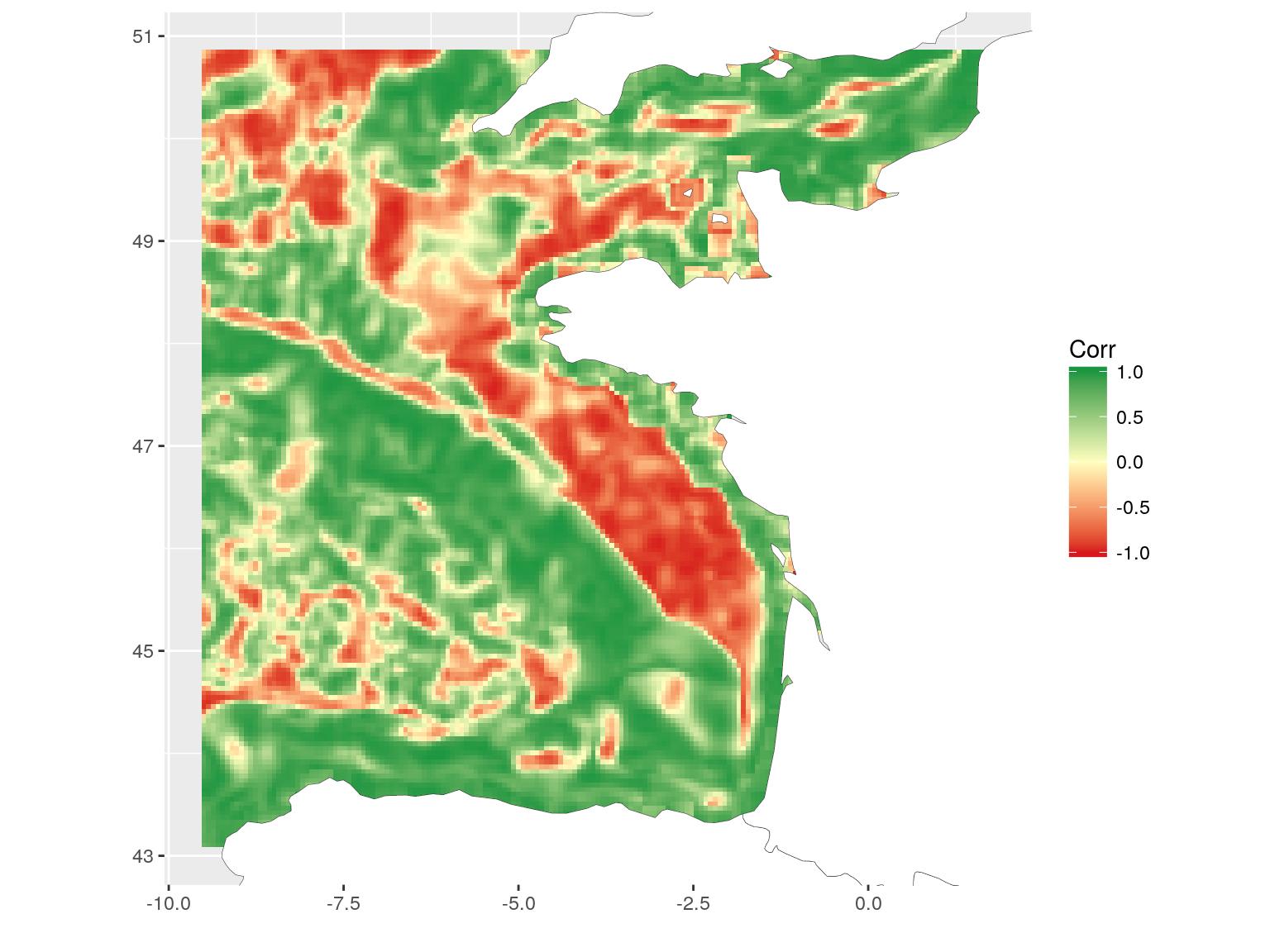
Récemment, j’ai utilisé cette fonction de corrélation locale pour un projet avec Marion (Marion Louveaux, bio-image analyste] et ça n’avait aucun rapport avec des données géographiques. Il s’agissait d’examiner la concentration des composants chimiques dans une racine de plante au cours du temps. Les données étaient stockées dans une matrice avec la position dans la racine en lignes et le temps en colonnes. La corrélation position-temps locale permet d’observer l’évolution des composants dans la racine.
Si vous ouvrez votre esprit, vous verrez que cette merveilleuse bibliothèque {raster} peut être utilisée à d’autres fins que les données géographiques…
Le script R de cet article est disponible sur mon dépôt Github ‘blog tips’
Citation :
Merci de citer ce travail avec :
Rochette Sébastien. (2018, janv.. 27). "Corrélation spatiale entre rasters". Retrieved from https://statnmap.com/fr/2018-01-27-correlation-spatiale-entre-rasters/.
Citation BibTex :
@misc{Roche2018Corré,
author = {Rochette Sébastien},
title = {Corrélation spatiale entre rasters},
url = {https://statnmap.com/fr/2018-01-27-correlation-spatiale-entre-rasters/},
year = {2018}
}