Il y a quelques semaines, Christina Buelow a demandé sur Twitter comment elle pouvait créer les polygones des eaux côtières en partant des polygones terrestres, et en s’assurant de ne pas avoir de chevauchement de zone tampon. Cela permettrait de savoir quel secteur terrestre est le plus proche de n’importe quel endroit dans la mer. Explorons quelle solution est ressortie des discussions.
Pas l’temps de tout lire !
- Installer le package {cartomisc} depuis GitHub
- Télécharger quelques données
# Define where to save the dataset
extraWD <- tempdir()
# Get some data available to anyone
if (!file.exists(file.path(extraWD, "departement.zip"))) {
githubURL <- "https://github.com/statnmap/blog_tips/raw/master/2018-07-14-introduction-to-mapping-with-sf-and-co/data/departement.zip"
download.file(githubURL, file.path(extraWD, "departement.zip"))
unzip(file.path(extraWD, "departement.zip"), exdir = extraWD)
}- Reduire le jeu de données à une région plus petite
library(sf)
# remotes::install_github("statnmap/cartomisc")
library(cartomisc)
library(dplyr)
library(ggplot2)
departements_l93 <- read_sf(dsn = extraWD, layer = "DEPARTEMENT")
# Reduce dataset
bretagne_l93 <- departements_l93 %>%
filter(NOM_REG == "BRETAGNE")- Calculer la zone tampon régionale avec
regional_seas()
bretagne_regional_2km_l93 <- regional_seas(
x = bretagne_l93,
group = "NOM_DEPT",
dist = units::set_units(30, km), # buffer distance
density = units::set_units(0.5, 1/km) # density of points (the higher, the more precise the region attribution)
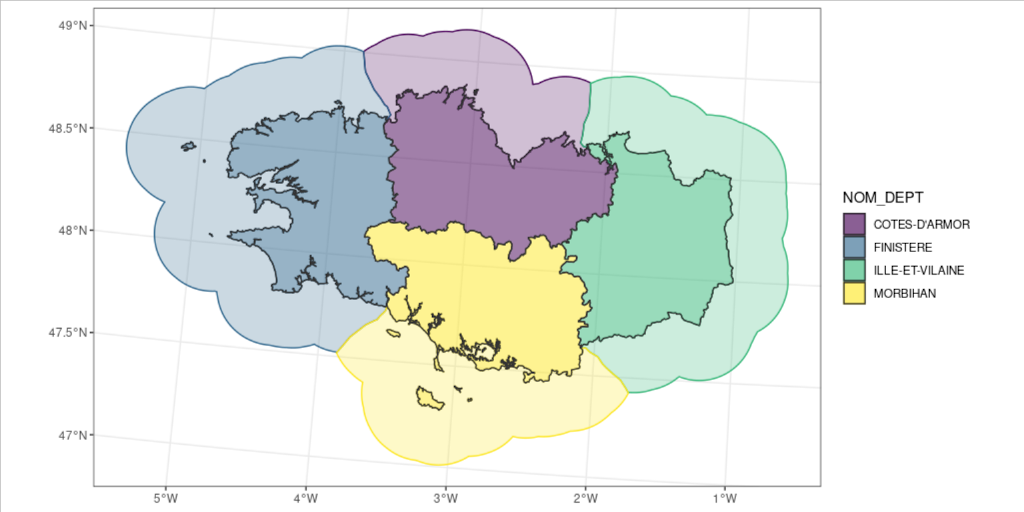
)- Afficher la carte des résultats
ggplot() +
geom_sf(data = bretagne_regional_2km_l93,
aes(colour = NOM_DEPT, fill = NOM_DEPT),
alpha = 0.25) +
geom_sf(data = bretagne_l93,
aes(fill = NOM_DEPT),
colour = "grey20",
alpha = 0.5) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
theme_bw()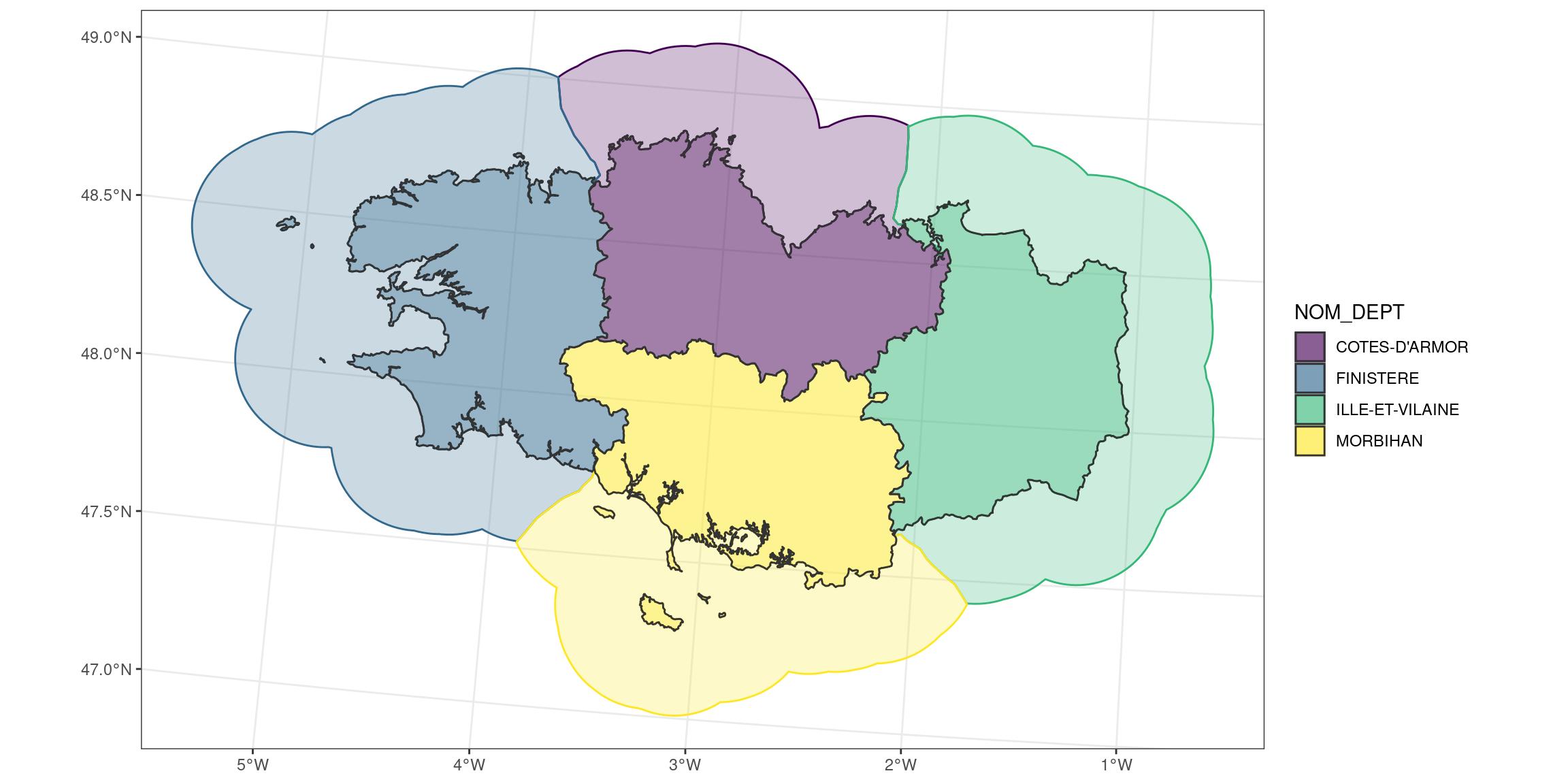
Si vous voulez connaître la version longue de cette histoire, vous pouvez continuer à lire, sinon, vous avez tout ce qu’il faut pour essayer sur vos données.
Créer une zone tampon autour des polygones adjacents
Comme toujours lorsqu’on essaie de trouver une solution à un problème spécifique, on commence par un exemple reproductible, communément appelé “reprex” chez les utilisateurs R. Cela signifie que nous créons le code minimal qui permet à quiconque d’exécuter le code, de faire face au même problème et de montrer tout ce que nous avons testé. Lors de la discussion sur Twitter, je n’ai pas eu assez de temps pour trouver une solution, mais j’ai préparé un gist avec mes essais, le point que j’avais atteint et, plus important encore, ce que je comptais réaliser après.
Tout d’abord, chargeons quelques packages
library(sf)
library(ggplot2)
library(dplyr)
my_blue <- "#1e73be"Ensuite, téléchargeons des données
# Define where to save the dataset
extraWD <- "."
# Get some data available to anyone
if (!file.exists(file.path(extraWD, "departement.zip"))) {
githubURL <- "https://github.com/statnmap/blog_tips/raw/master/2018-07-14-introduction-to-mapping-with-sf-and-co/data/departement.zip"
download.file(githubURL, file.path(extraWD, "departement.zip"))
unzip(file.path(extraWD, "departement.zip"), exdir = extraWD)
}Je réduis le jeu de données pour mieux voir ce que je fais. Ici, je filtre sur la région Bretagne.
Comme vous pouvez le voir, il y a 4 départements dans cette région, donc 4 entités spatiales.
departements_l93 <- read_sf(dsn = extraWD, layer = "DEPARTEMENT")
# Reduce dataset
bretagne_l93 <- departements_l93 %>%
filter(NOM_REG == "BRETAGNE")
bretagne_l93## Simple feature collection with 4 features and 11 fields
## Geometry type: MULTIPOLYGON
## Dimension: XY
## Bounding box: xmin: 99217.1 ymin: 6704195 xmax: 400572.3 ymax: 6881964
## Projected CRS: RGF93_Lambert_93
## # A tibble: 4 x 12
## ID_GEOFLA CODE_DEPT NOM_DEPT CODE_CHF NOM_CHF X_CHF_LIEU Y_CHF_LIEU X_CENTROID
## * <chr> <chr> <chr> <chr> <chr> <int> <int> <int>
## 1 DEPARTEM… 35 ILLE-ET… 238 RENNES 351870 6789646 326237
## 2 DEPARTEM… 22 COTES-D… 278 SAINT-… 274947 6839206 260216
## 3 DEPARTEM… 29 FINISTE… 232 QUIMPER 171326 6789950 115669
## 4 DEPARTEM… 56 MORBIHAN 260 VANNES 267889 6744074 256790
## # … with 4 more variables: Y_CENTROID <int>, CODE_REG <chr>, NOM_REG <chr>,
## # geometry <MULTIPOLYGON [m]>Maintenant, je peux créer un graphique avec une zone tampon autour des polygones séparés.
# Create buffer
bretagne_buffer_l93 <- bretagne_l93 %>%
st_buffer(
dist =
units::set_units(10, km)
)
ggplot() +
geom_sf(data = bretagne_buffer_l93, fill = my_blue, alpha = 0.2, colour = my_blue) +
geom_sf(data = bretagne_l93, fill = NA) +
theme_bw()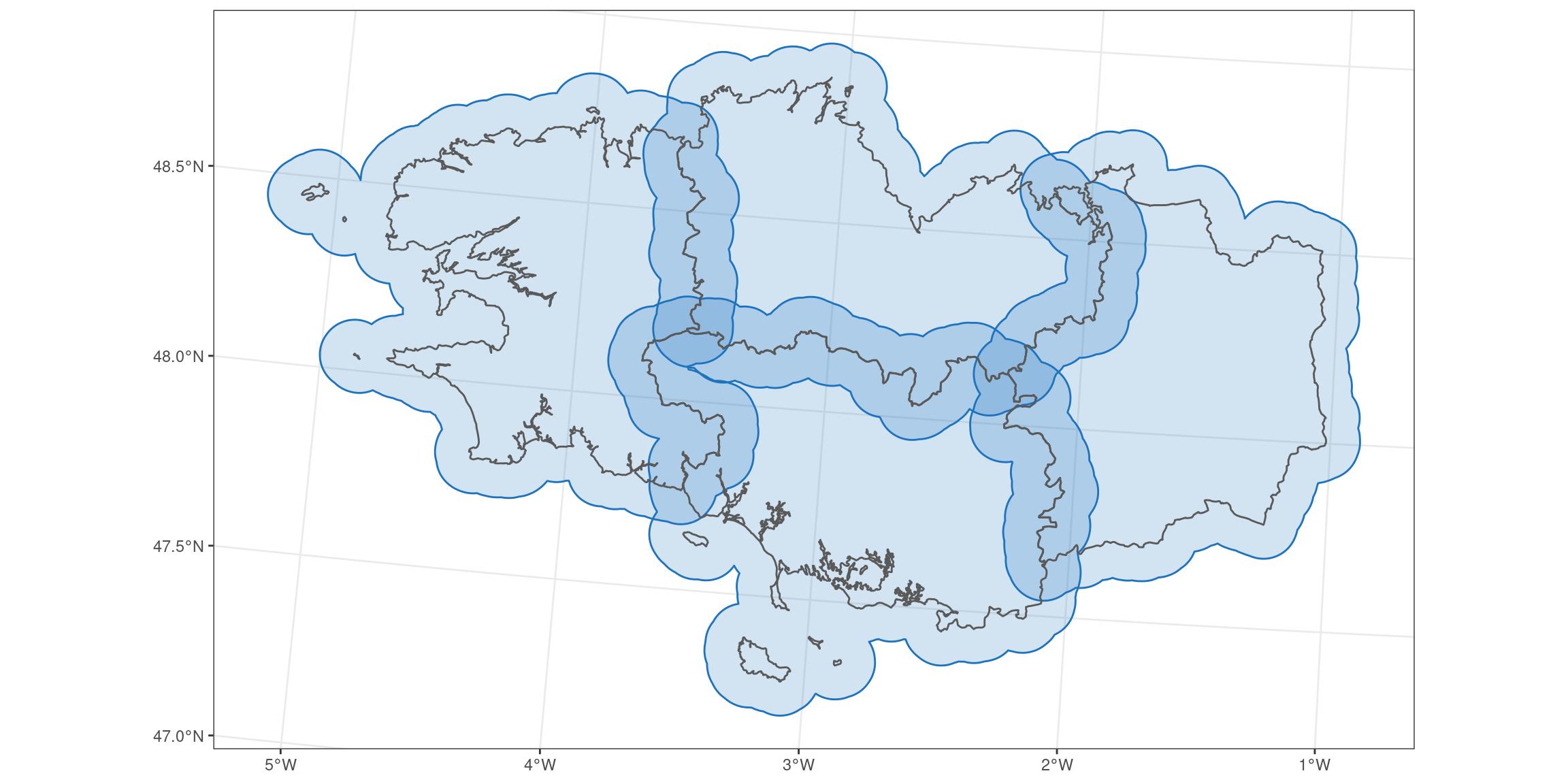
Vous pouvez voir qu’il y a des zones de chevauchement.
C’est là que les polygones de Voronoï vont venir nous aider. À partir d’ici, je vais prendre quelques idées que Christina a recueillies lors de ses discussions sur Twitter. Son code pour les zones tampons des zones côtières est ici.
# Create a merged region entity
bretagne_union_l93 <- bretagne_l93 %>%
summarise()
ggplot() +
geom_sf(data = bretagne_union_l93, colour = my_blue, fill = NA)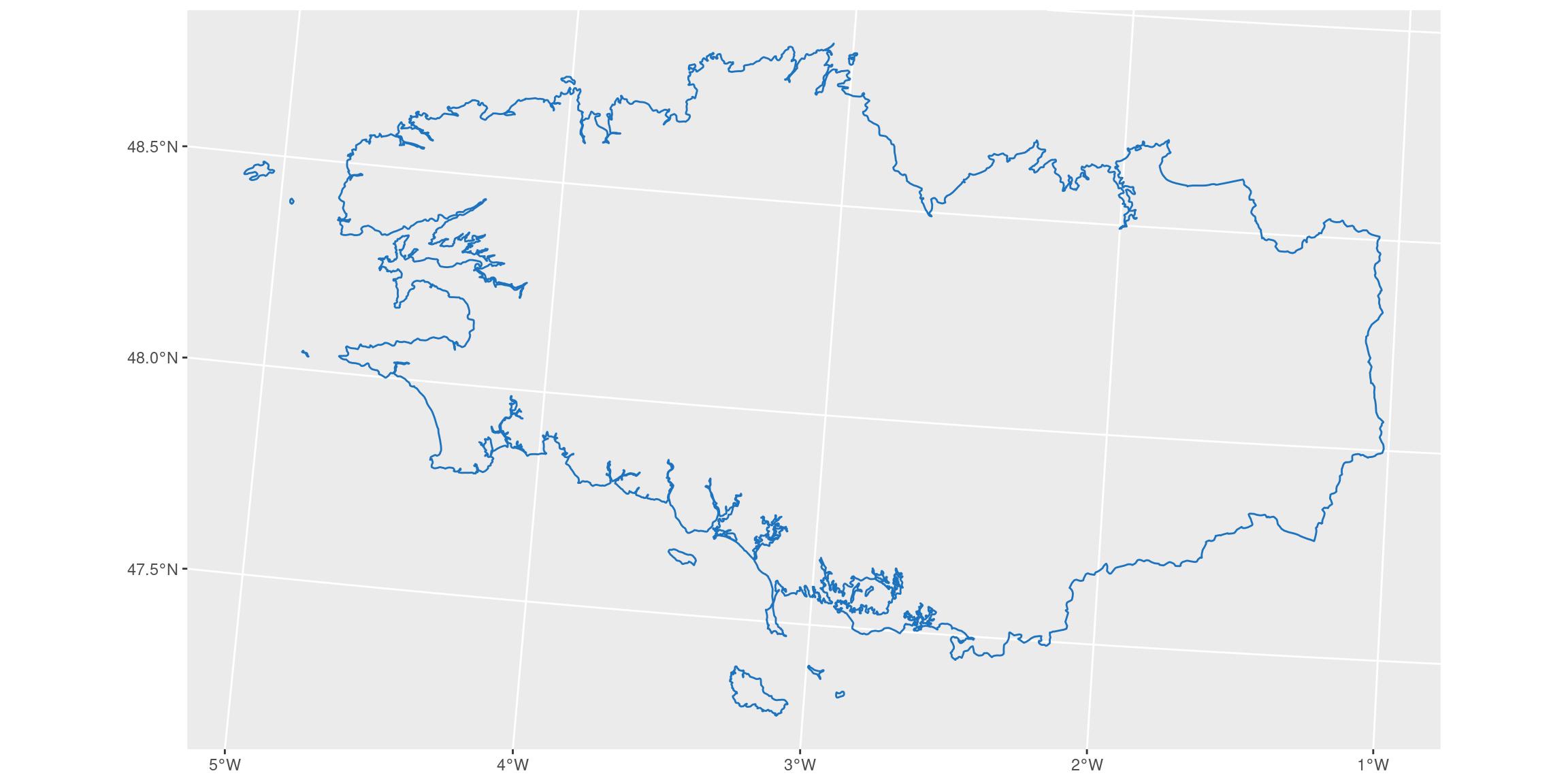
# Create a doughnut for regional seas areas, 30km off coasts
bretagne_seas_l93 <- bretagne_union_l93 %>%
st_buffer(
dist = units::set_units(30, km)
) %>%
st_cast()
ggplot() +
geom_sf(data = bretagne_seas_l93, colour = my_blue, fill = NA)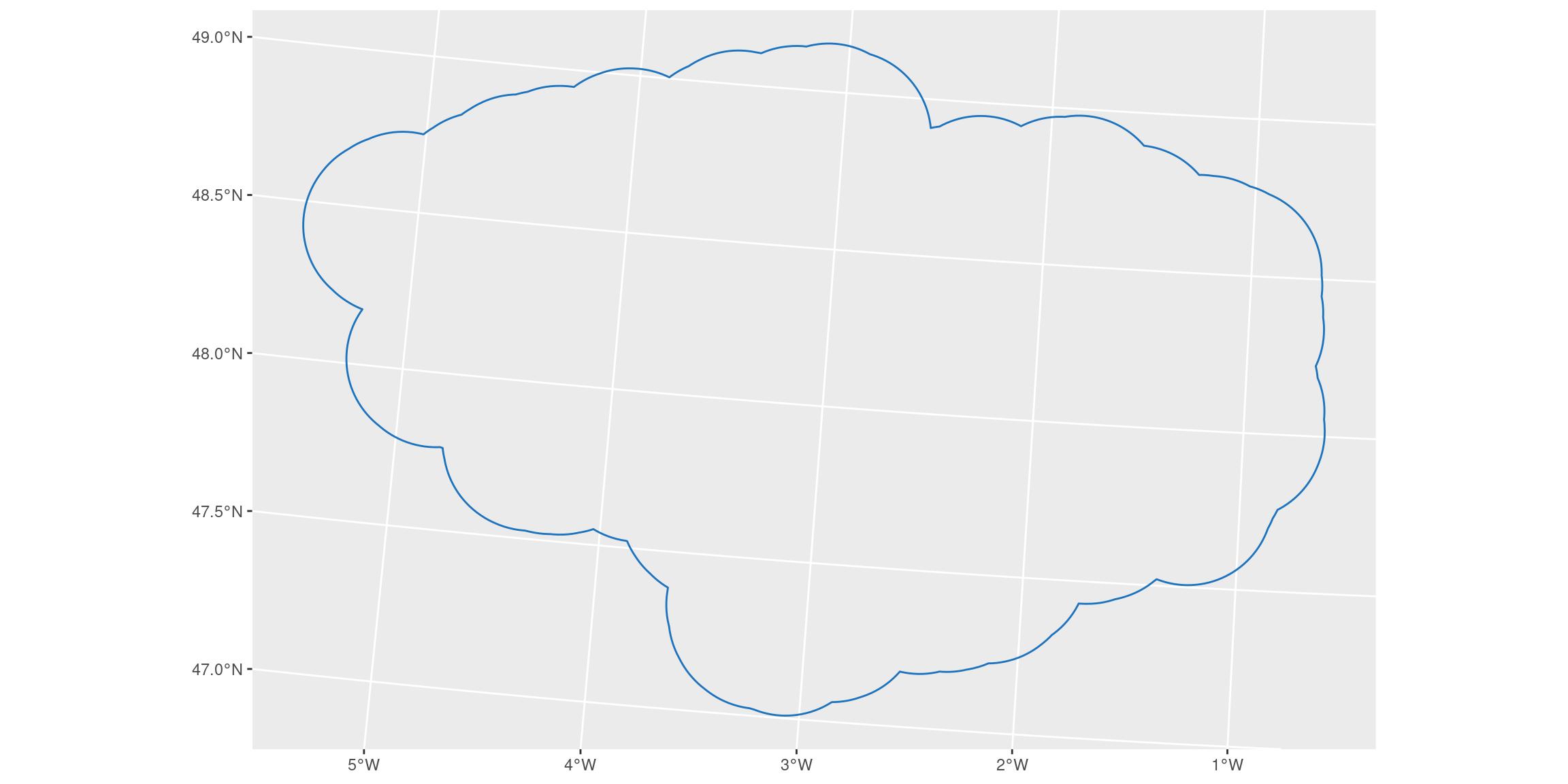
# Remove inside terrestrial parts
bretagne_donut_l93 <- bretagne_seas_l93 %>%
st_difference(bretagne_union_l93) %>%
st_cast()
ggplot() +
geom_sf(data = bretagne_donut_l93, colour = my_blue, fill = my_blue, alpha = 0.2)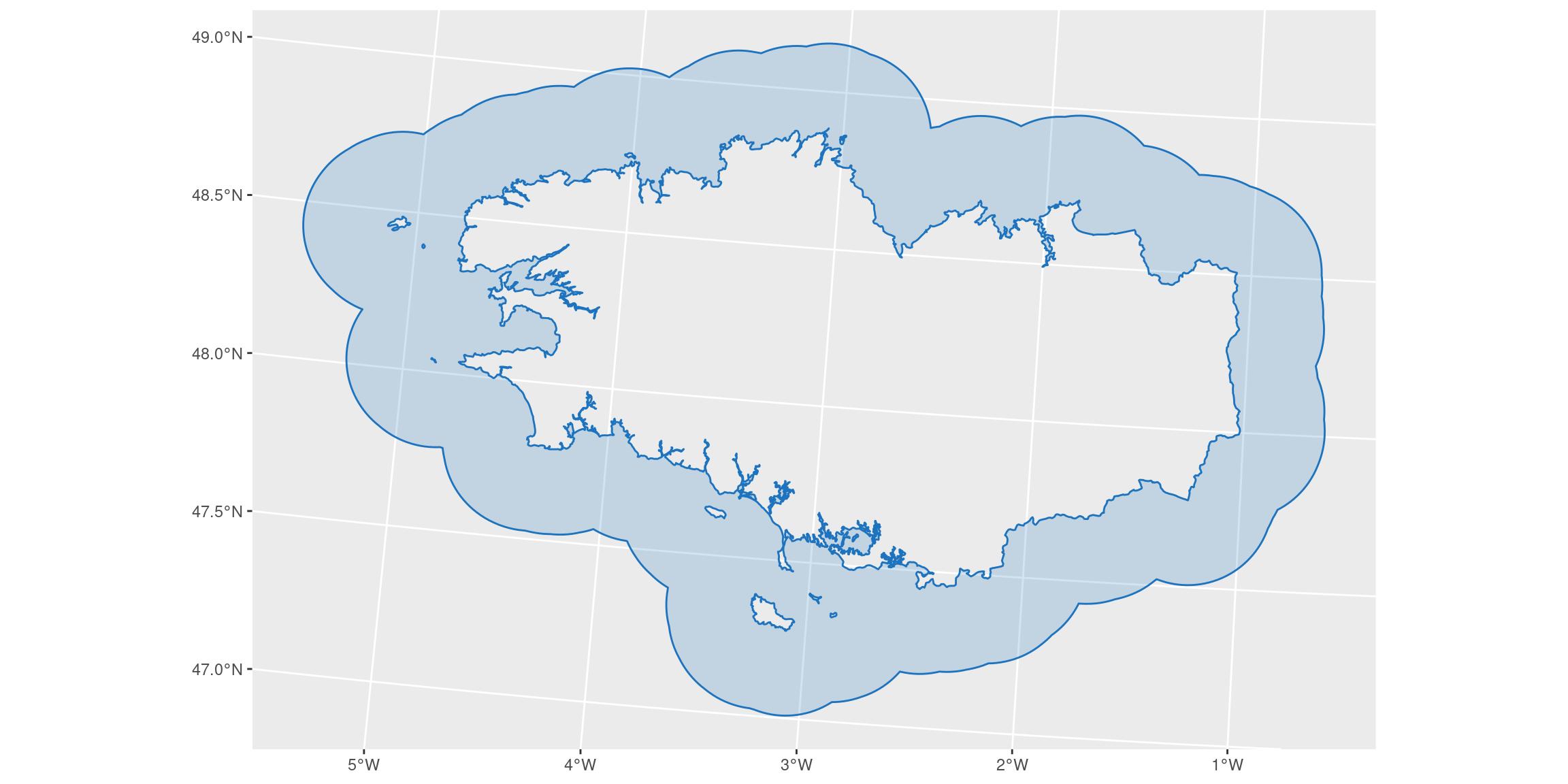
Divisons cette zone en plusieurs polygones
Nous utilisons le trait de côte comme base.
# density <- units::set_units(0.1, 1/km)
# First merge everything and transform as lines
bretagne_points_l93 <- bretagne_union_l93 %>%
# transform polygons as multi-lines
st_cast("MULTILINESTRING") %>%
# transform multi-lines as separate unique lines
st_cast("LINESTRING") %>%
# transform as points, 0.1 per km (= 1 every 10 km)
# Choose density according to precision needed
st_line_sample(density = units::set_units(0.1, 1/km)) %>%
st_sf() #%>%
# remove empty geometry (small islands where sample is not possible with this density)
# filter(!st_is_empty(geometry))
ggplot() +
geom_sf(data = bretagne_points_l93, colour = my_blue, fill = NA)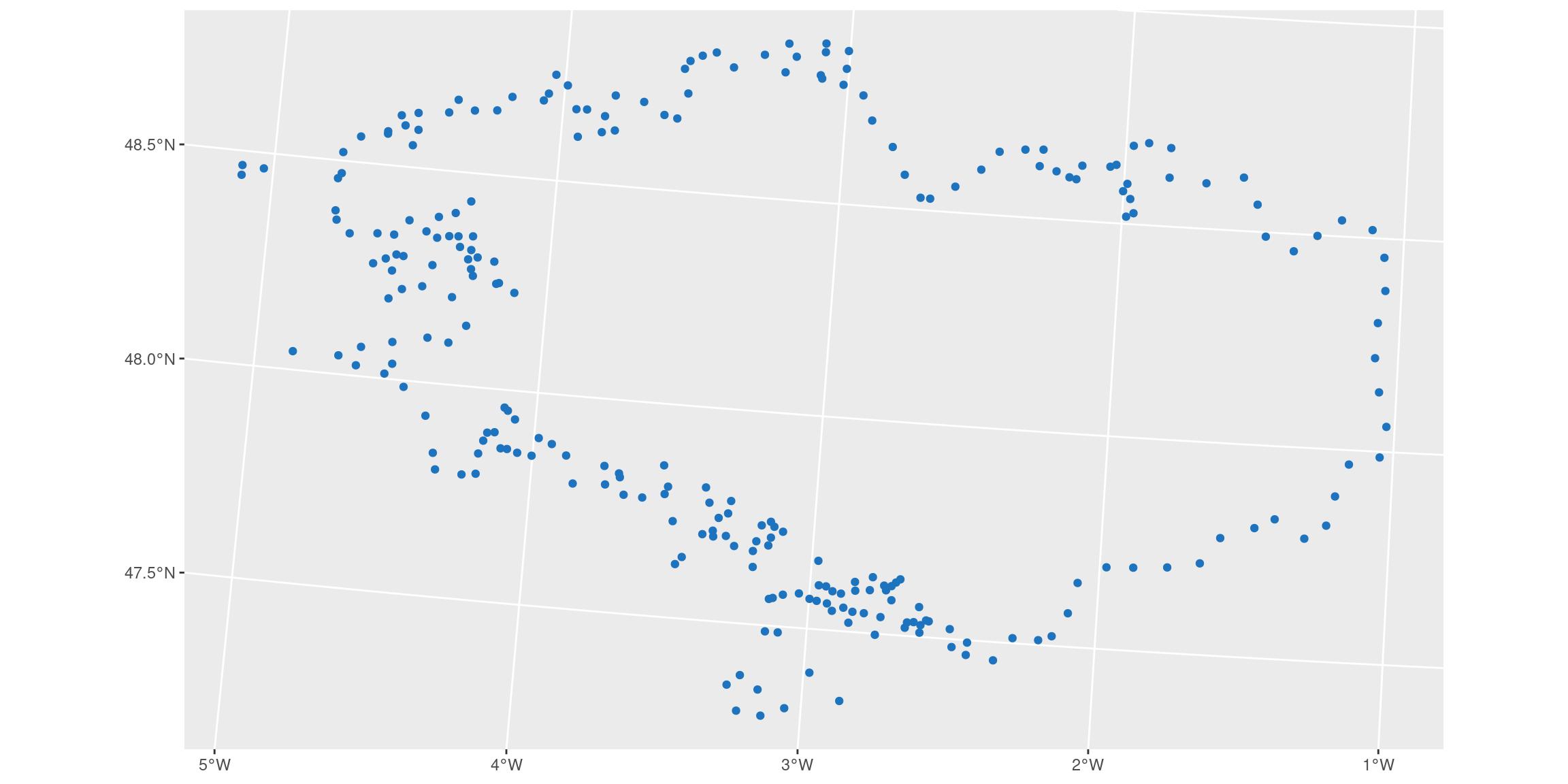
# Create voronoi polygons around points
bretagne_voronoi_l93 <- bretagne_points_l93 %>%
st_combine() %>%
st_voronoi() %>%
st_cast()
ggplot() +
geom_sf(data = bretagne_voronoi_l93, colour = my_blue, fill = NA)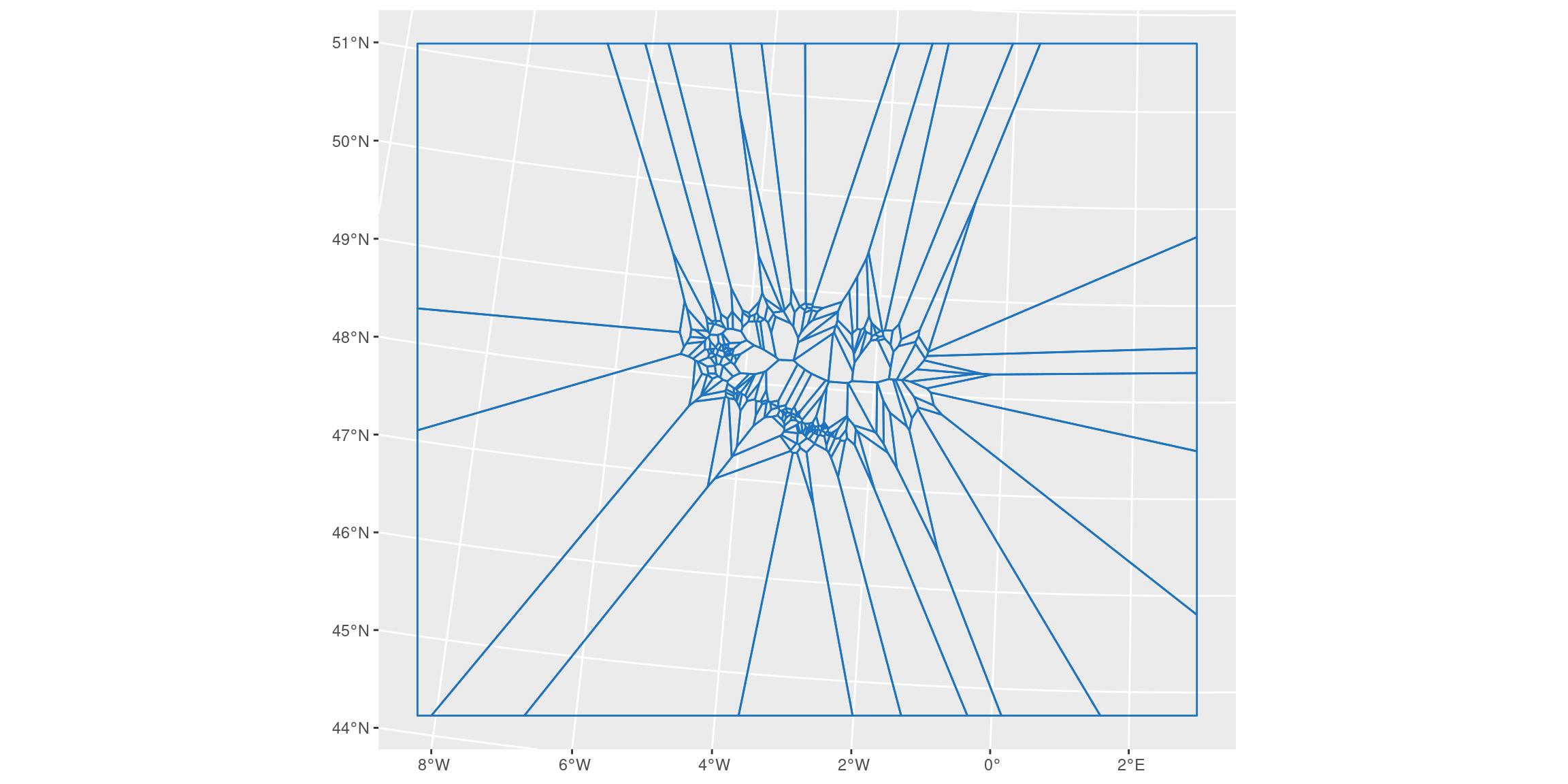
Jointure des polygones de voronoi et des polygones type “doughnut”
bretagne_voronoi_donut_l93 <- bretagne_voronoi_l93 %>%
st_intersection(bretagne_donut_l93) %>%
st_cast() %>%
st_sf()
ggplot() +
geom_sf(data = bretagne_voronoi_donut_l93, colour = my_blue, fill = NA)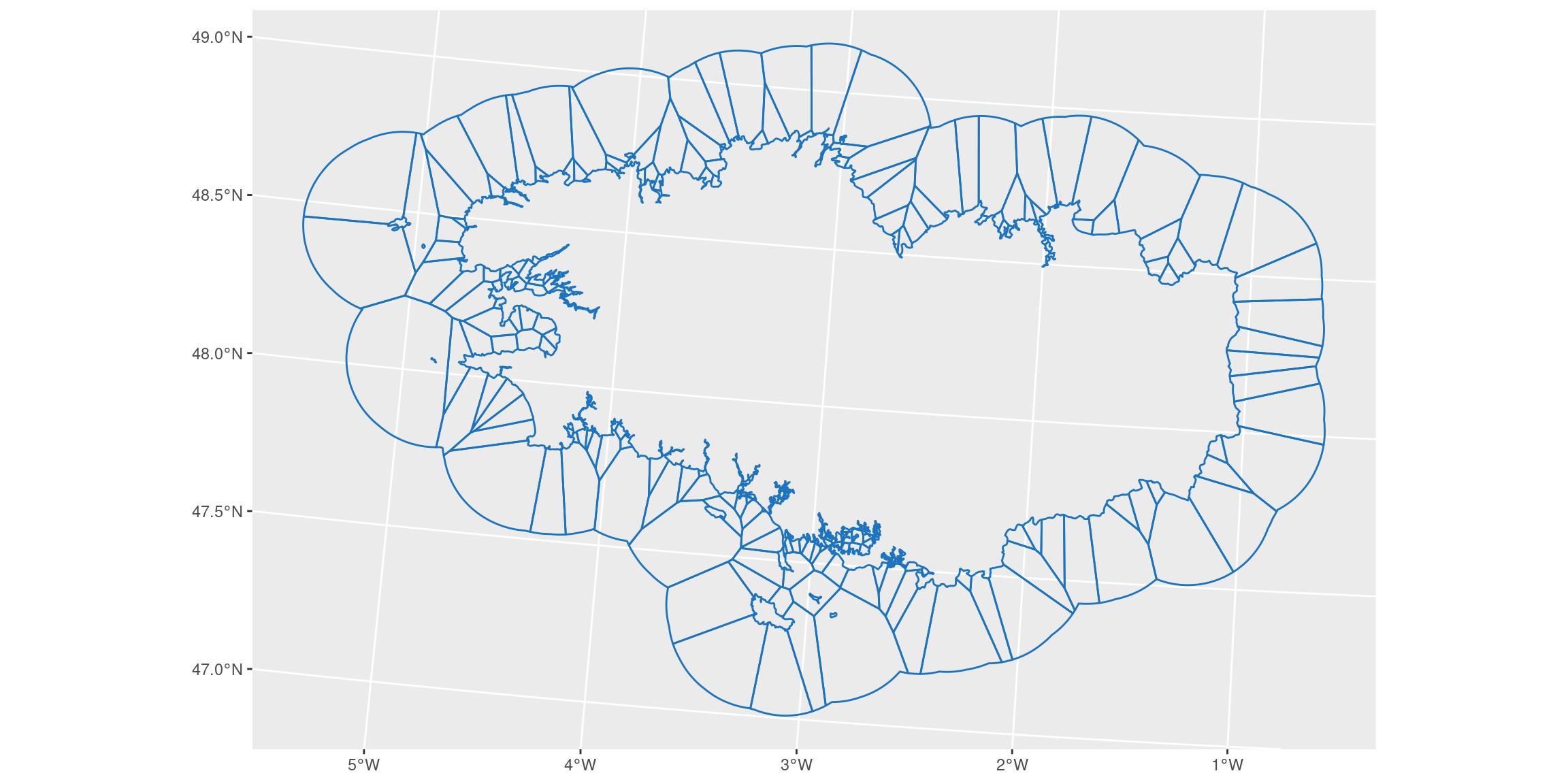
Joindre les polygones de voronoi-donut au jeu de données original pour l’intersection avec les régions
Nous réalisons une sorte de left_join (jointure à gauche) basée sur des attributs spatiaux en utilisant st_join(). Cela permettra de conserver la géométrie originale de nos polygones de Voronoï, tout en récupérant les variables du jeu de données terrestres jointes lorsqu’il y a intersection.
# Equivalent to left_join
bretagne_vd_region_l93 <- bretagne_voronoi_donut_l93 %>%
st_join(bretagne_l93)
ggplot() +
geom_sf(data = bretagne_vd_region_l93,
aes(colour = NOM_DEPT, fill = NOM_DEPT),
alpha = 0.25)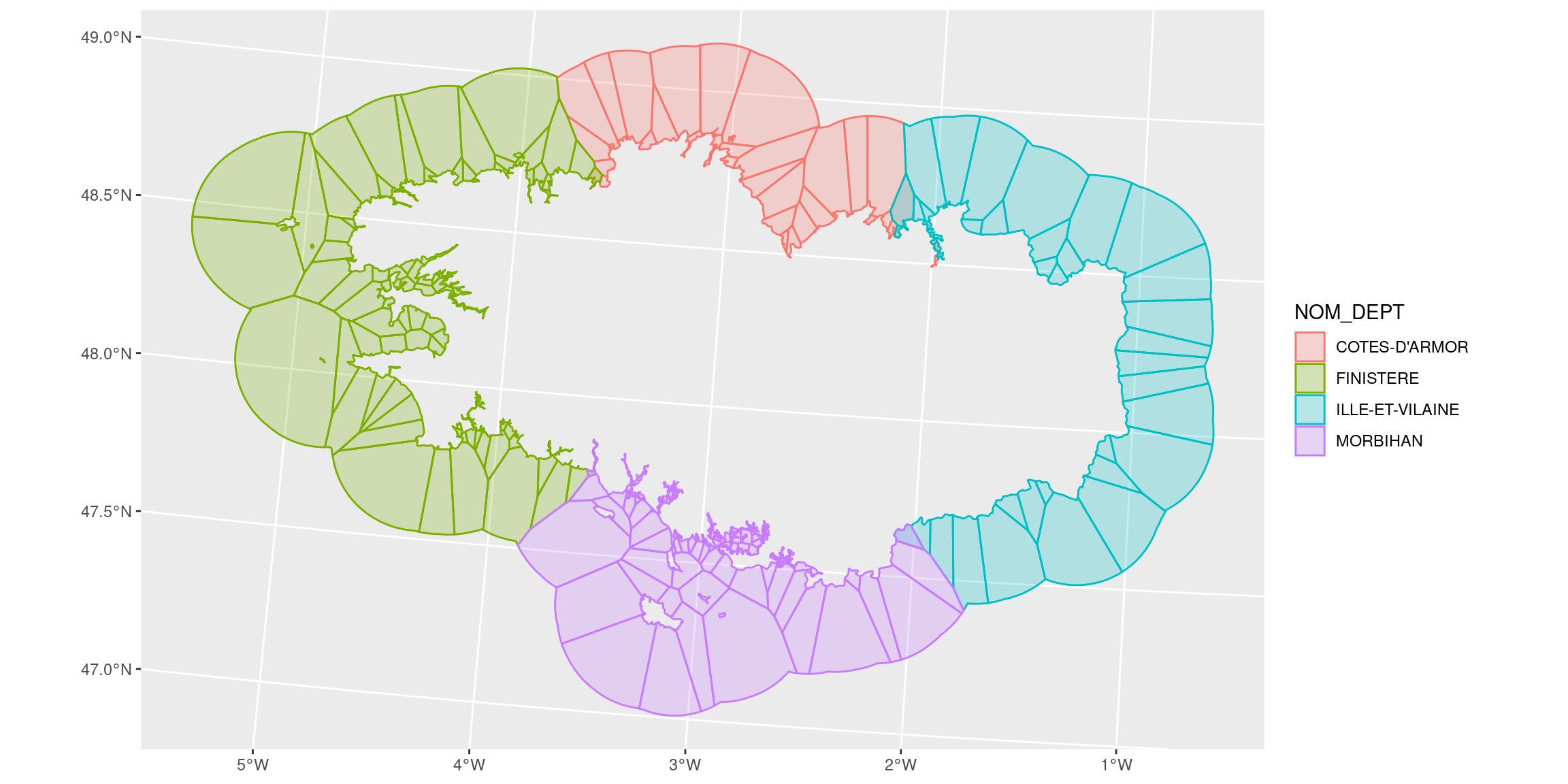
# Interactive view
mapview::mapview(bretagne_vd_region_l93, zcol = "NOM_DEPT")Si on zoome sur la carte en utilisant mapview(), vous pouvez voir que les polygones entre deux régions connaissent une double attribution. C’est parce que les polygones de Voronoï se joignent avec les deux régions. Si vous voulez réduire la taille des polygones qui se chevauchent, vous pouvez augmenter la densité des points utilisée dans st_line_sample() pour créer les polygones de voronoï. Cependant, nous aurons toujours la double attribution. Rappelez-vous que st_join() fonctionne comme left_join(). S’il y a des clés de jointure en double, les entités sont doublées.
Pour éviter cela, nous devrons choisir entre l’une ou l’autre des régions. D’abord, nous donnons un numéro aux polygones de Voronoï avant de les joindre pour pouvoir identifier ceux qui sont doublés. Ensuite, nous choisirons de ne garder que le premier d’entre eux dans la liste. Notez que summarise_all n’existe pas pour les objets {sf}. Je ne peux donc pas utiliser group_by(id_v) + summarise_all(first) comme pour les jeux de données classiques, ni la nouvelle fonction across(). J’utilise distinct() avec le paramètre keep_all = TRUE pour qu’il conserve toutes les variables avec la première entité pour chaque polygone de Voronoï.
bretagne_vd_region_distinct_l93 <- bretagne_voronoi_donut_l93 %>%
mutate(id_v = 1:n()) %>%
st_join(bretagne_l93) %>%
# group_by(id_v) %>%
# summarise(across(.cols = everything(), .fns = first)) # Not on {sf}
# summarise_all(.funs = first) # Not on {sf}
distinct(id_v, .keep_all = TRUE)
ggplot() +
geom_sf(data = bretagne_vd_region_distinct_l93,
aes(colour = NOM_DEPT, fill = NOM_DEPT),
alpha = 0.25)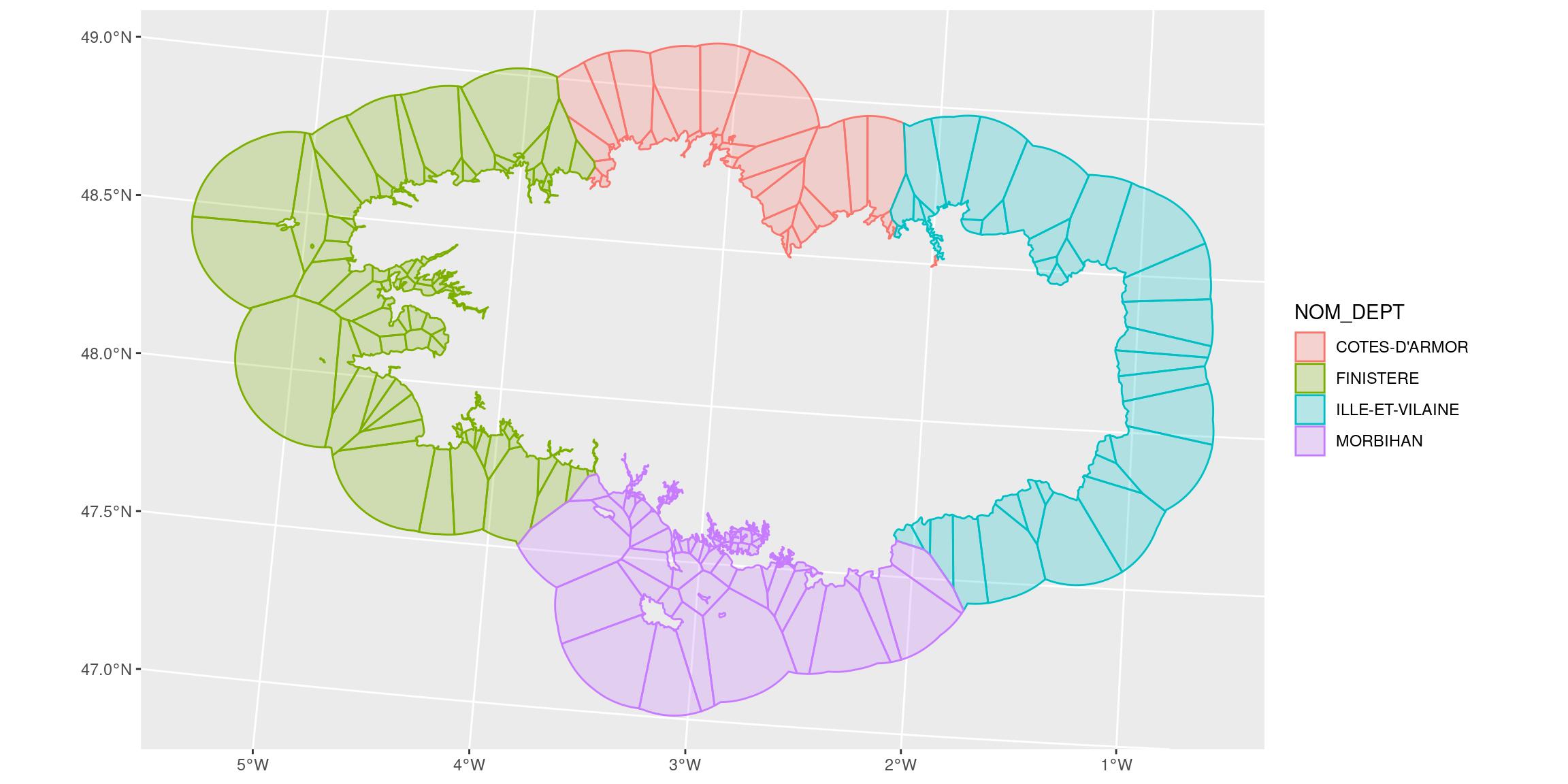
# Interactive view
mapview::mapview(bretagne_vd_region_distinct_l93, zcol = "NOM_DEPT")Jointure de tous les polygones en fonction de la variable d’intérêt
Je peux maintenant faire l’union des polygones.
bretagne_seas_l93 <- bretagne_vd_region_distinct_l93 %>%
group_by(NOM_DEPT) %>%
summarise()
ggplot() +
geom_sf(data = bretagne_seas_l93,
aes(colour = NOM_DEPT, fill = NOM_DEPT),
alpha = 0.25) +
geom_sf(data = bretagne_l93,
aes(fill = NOM_DEPT),
colour = "grey20",
alpha = 0.5)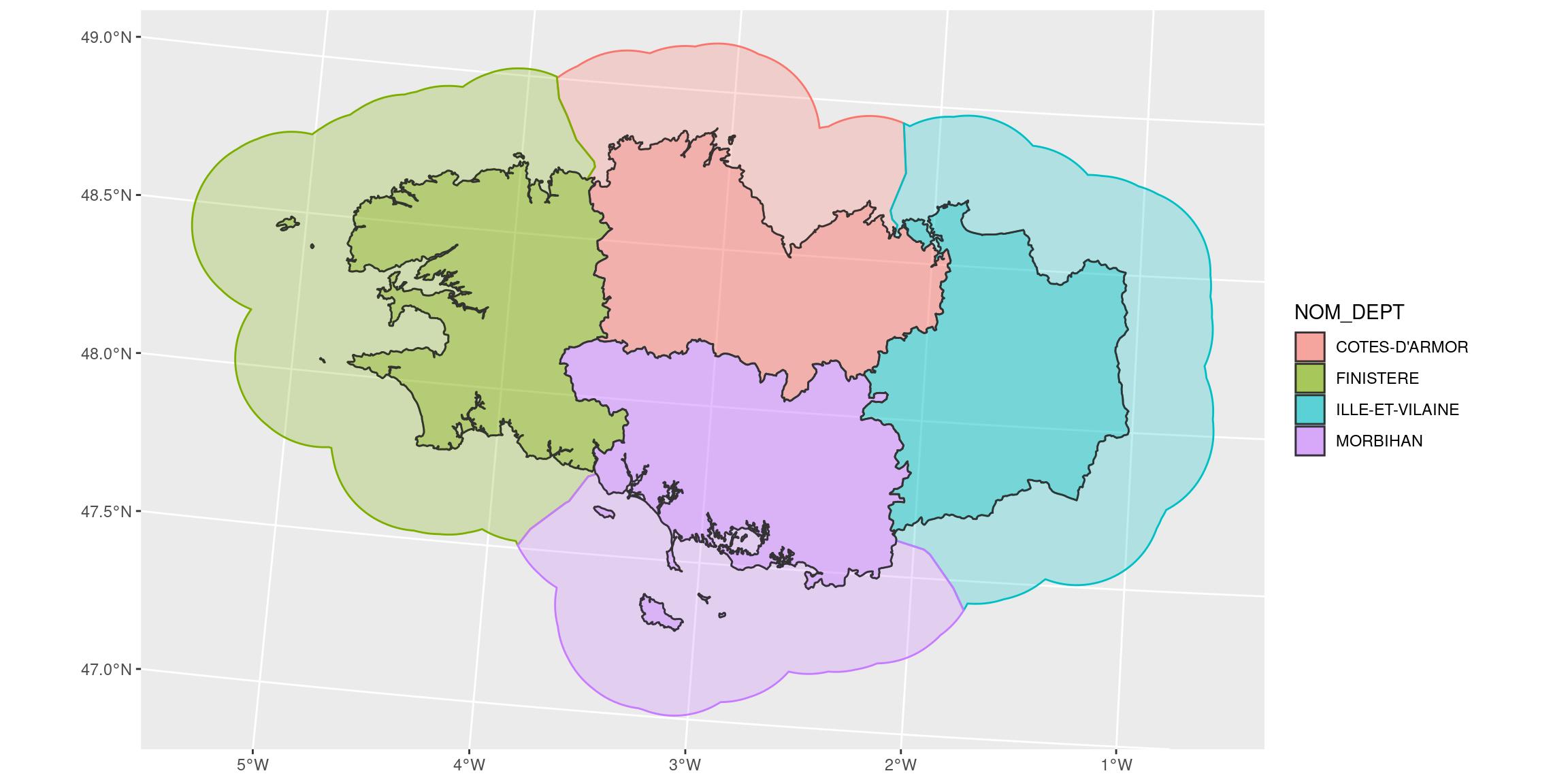
Créer des zones tampons avec l’attribut de la région la plus proche avec {cartomisc}
Je ne peux pas vous laisser partir sans une fonction qui rassemble tout et son application avec une plus grande densité de points le long des côtes. La fonction est écrite ci-dessous, mais elle est également disponible avec mon package {cartomisc} : “Divers outils pour la manipulation et l’analyse de données spatiales”
#' @param x Spatial polygon layer
#' @param group Character. The grouping variable for your subareas
#' @param dist distance from coasts of the buffer area. See ?sf::st_buffer
#' @param density density of points along the coastline
#'
#' @importFrom sf st_buffer st_cast st_difference st_line_sample st_sf st_geometry
#' @importFrom sf st_combine st_voronoi st_intersection st_join
#' @importFrom dplyr summarise mutate distinct group_by_at
regional_seas <- function(x,
group,
dist = units::set_units(30, km),
density = units::set_units(0.1, 1/km)
) {
# Create a merged region entity
x_union <- x %>%
summarise()
# Create a doughnut for regional seas areas, 30km off coasts
x_donut <- x_union %>%
st_buffer(
dist = dist
) %>%
st_cast() %>%
# Remove inside terrestrial parts
st_difference(x_union) %>%
st_cast()
# First merge everything and transform as lines
x_lines <- x_union %>%
# transform polygons as multi-lines
st_cast("MULTILINESTRING") %>%
# transform multi-lines as separate unique lines
st_cast("LINESTRING")
# Then as regular points
x_points <- x_lines %>%
# transform as points, 0.1 per km (= 1 every 10 km)
# Choose density according to precision needed
st_line_sample(density = density) %>%
st_sf() #%>%
# remove empty geometry (small islands where sample is not possible with this density)
# filter(!st_is_empty(geometry))
if (any(st_is_empty(x_points$geometry))) {
# Add original islands if empty
x_lines_multipoints <- x_lines %>%
st_cast("MULTIPOINT")
# replace geometry with original lines as points
st_geometry(x_points)[st_is_empty(x_points$geometry)] <-
st_geometry(x_lines_multipoints)[st_is_empty(x_points$geometry)]
x_points <- x_points %>% st_cast()
warning("There were empty geometries after sampling points along coastlines. ",
"'density' was probably not big enough for some isolated polygons. ",
"They have been reinstated with their original resolution")
}
# Create voronoi polygons around points
x_vd_region_distinct <- x_points %>%
st_combine() %>%
st_voronoi() %>%
st_cast() %>%
st_intersection(x_donut) %>%
st_cast() %>%
st_sf() %>%
mutate(id_v = 1:n()) %>%
st_join(x) %>%
# group_by(id_v) %>%
# summarise(across(.cols = everything(), .fns = first)) # Not on {sf}
# summarise_all(.funs = first) # Not on {sf}
distinct(id_v, .keep_all = TRUE)
# group by variable
if (!missing(group)) {
x_seas <- x_vd_region_distinct %>%
group_by_at(group) %>%
summarise()
return(x_seas)
}
return(x_vd_region_distinct)
}Appliquer la fonction à notre région avec une résolution de 2 km (densité = 0,5 points par km)
# remotes::install_github("statnmap/cartomisc")
library(cartomisc)
bretagne_regional_2km_l93 <- regional_seas(
x = bretagne_l93,
group = "NOM_DEPT",
density = units::set_units(0.5, 1/km)
)
# Plot
ggplot() +
geom_sf(data = bretagne_regional_2km_l93,
aes(colour = NOM_DEPT, fill = NOM_DEPT),
alpha = 0.25) +
geom_sf(data = bretagne_l93,
aes(fill = NOM_DEPT),
colour = "grey20",
alpha = 0.5) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
theme_bw()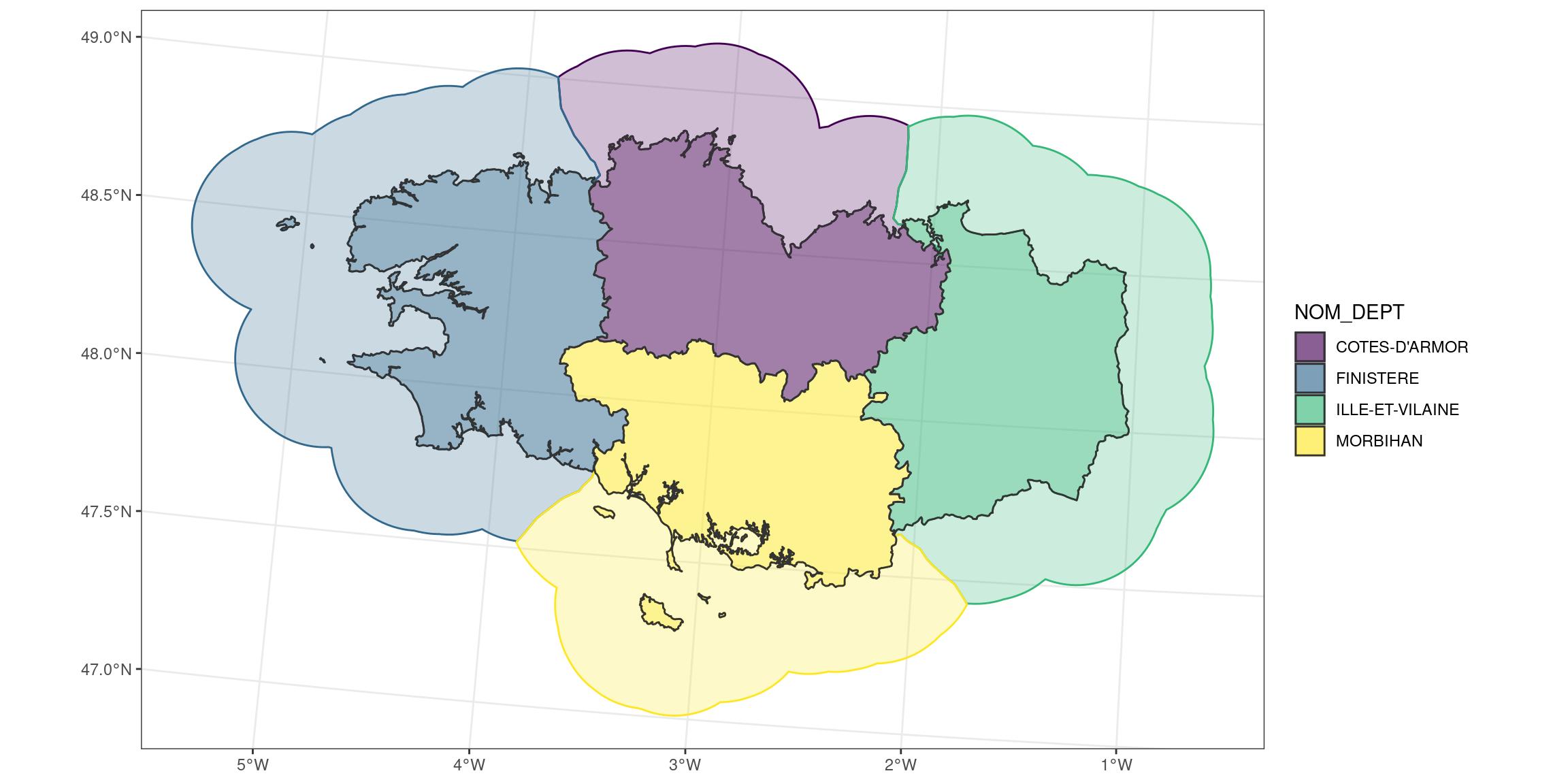
Test de {cartomisc}
Vous pouvez essayer cette fonction disponible sur Github dans le package {cartomisc} avec vos propres jeux de données. Si vous souhaitez l’améliorer ou si vous rencontrez des problèmes avec des données spatiales spécifiques, n’hésitez pas à ouvrir une pull request ou une issue.
Comme toujours, le code et les données sont disponibles sur https://github.com/statnmap/blog_tips
Citation :
Merci de citer ce travail avec :
Rochette Sébastien. (2020, août. 19). "Zones tampons avec attributs du plus proche voisin". Retrieved from https://statnmap.com/fr/2020-08-13-zones-tampons-avec-attributs-du-plus-proche-voisin/.
Citation BibTex :
@misc{Roche2020Zones,
author = {Rochette Sébastien},
title = {Zones tampons avec attributs du plus proche voisin},
url = {https://statnmap.com/fr/2020-08-13-zones-tampons-avec-attributs-du-plus-proche-voisin/},
year = {2020}
}