Jouons avec les outils de cartographie sur des données non géographiques ! {rayshader} est cool sur les raster, mais pourquoi ne pas l’utiliser sur des cellules végétales segmentées ?
Utilisation des outils de cartographie hors de leur contexte
Ce n’est pas la première fois que j’utilise des outils de cartographie pour faire autre chose que de la cartographie. Dans mon article de blog Une application Shiny pour la comparaison experte d’images, j’ai parlé d’un article écrit avec Marion Louveaux, une analyste de données issues d’images biologiques travaillant sur les images de microscopie de tissus végétaux. Dans cet article scientifique, nous avons utilisé des outils pour mesurer l’autocorrélation spatiale de l’intensité des couleurs des pixels d’images obtenues au microscope confocal.
Notre collaboration continue à travers différents packages R, en développement sur le compte github de Marion. Dans cet article de blog, j’utiliserai les sorties d’images 3D issues d’une segmentation cellulaire réalisées par Marion avec MorphoGraphX software et importées sous forme de mesh 3D dans R grâce au package {mgx2r} (sa page de documentation {pkgdown} est ici).
Je voulais trouver une bonne raison de jouer avec les packages de Tyler Morgan-Wall pour la visualisation 3D de données raster : {rayshader} et {rayfocus}. Utiliser des images de segmentation de cellules en combinaison avec {rayshader}, c’est le défi du jour !
library(mgx2r)
library(rgl)
library(dplyr)
library(sf)
library(ggplot2)
library(raster)
library(rayshader)
library(magick)
# extraWD <- tempdir()Récupérer les données de segmentation cellulaire
Nous utilisons les données d’exemple de la vignette de {mgx2r}. Ce jeu de données est issu de MorphoGraphX. C’est une segmentation 3D surfacique d’une image 3D. Il est lu et transformé en mesh avec {mgx2r} pour pouvoir être utilisé avec {rgl}.
Par ailleurs, j’aime beaucoup les nouvelles fonctions ajoutées à {rgl}, comme ce rglwidget pour inclure des sorties {rgl} dans les documents HTML Rmarkdown ou dans les applications Shiny.
Cette image est un widget rgl interactif
filePly <- system.file("extdata", "full/mesh/mesh_meristem_full_T0.ply", package = "mgx2r")
myMesh <- read_mgxPly(
file = filePly, ShowSpecimen = FALSE, addNormals = TRUE,
MatCol = 1, header_max = 30)## [1] "Object has 7763 faces and 4158 vertices."try(rgl.close(), silent = TRUE)
plot3d(myMesh)
aspect3d("iso")
# Change view
view3d(theta = 30, phi = -40, zoom = 0.5)
rglwidget()Projection et transformation comme objet spatial
{rayshader} nécessite une matrice d’élévation mais ici nous avons un mesh 3D. Nous devons projeter le mesh en 2 dimensions et garder la troisième comme l’élévation. Par chance, le mesh est en 3D surfacique, ce qui signifie que sa projection peut être réalisée le long de l’axe Z. Pour d’autres types de mesh 3D, j’utiliserais transform3d avec une matrice de transformation personnalisée. La sélection du “z” pour la projection sera un peu délicate. Je ne vais pas aller plus loin sur ce sujet pour l’instant…
Ici, nous récupérons les coordonnées x/yen tant que projection et utilisons z comme la hauteur. Ces données sont transformées en objet spatial avec un système de coordonnées en mètres avec {sf}. Si vous cherchez à vous former à {sf}, vous pouvez commencer par mon précédent article “Initiation à la cartographie avec {sf} & Co.”.
mesh_sf <- t(myMesh$vb) %>%
as_tibble() %>%
st_as_sf(coords = c("x", "y"), crs = 2154)
ggplot(mesh_sf) +
geom_sf(aes(colour = z)) +
coord_sf(crs = 2154, datum = 2154) +
scale_colour_gradient2(low = "#d7191c", high = "#1a9641", mid = "#ffffbf",
midpoint = mean(mesh_sf$z))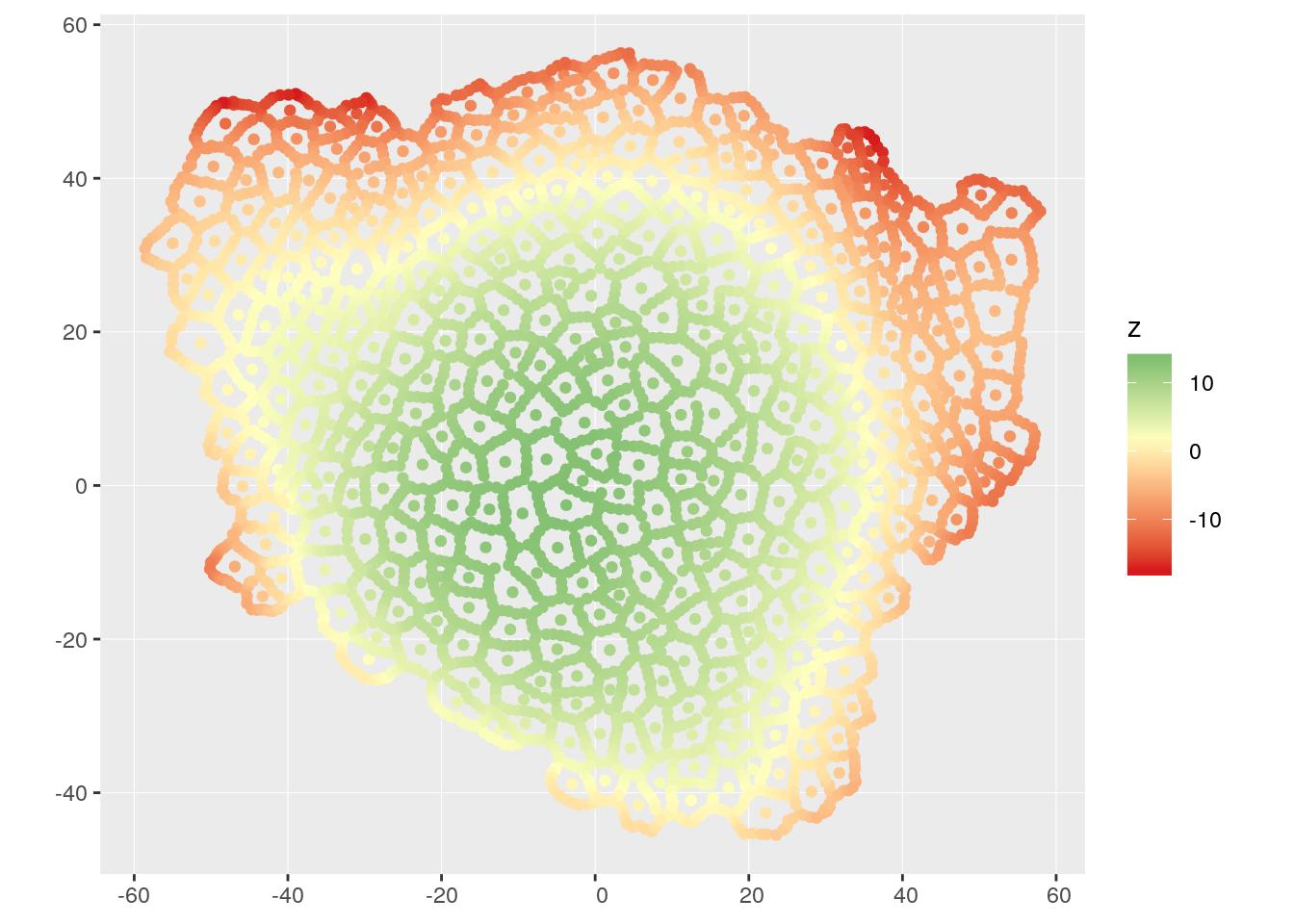
Interpolation sur un raster
Notre mesh transformé en objet spatial est un ensemble de données ponctuelles, mais {rayshader} a besoin un objet matriciel. Nous pouvons interpoler le mesh sur un raster avec un calcul de distance inverse comme décrit dans mon article de blog “interpolation-spatiale-sur-le-globe-terrestre-3D”.
Nous avons besoin de quelques étapes :
- Créer un raster vide avec les dimensions du mesh
- Transformer le raster en points avec {sf}
- Calculer la distance entre les points du mesh et du raster
- Faire l’interpolation avec inverse distance
- Les valeurs qui sont loin des données sont fixées à la valeur la plus basse (Ça évite les
NApour {rayshader}) - Remplir le raster vide avec les prédictions
# Create an empty raster with dimensions of the mesh ----
r <- raster(
as(mesh_sf, "Spatial"),
nrows = 100, ncols = 100,
crs = st_crs(mesh_sf)$proj4string
)
# Transform raster as spatial points with sf
r_sf <- st_as_sf(as.data.frame(coordinates(r)),
coords = c("x", "y"),
crs = 2154)
# Distance between points and raster ----
obs.r.dists <- st_distance(mesh_sf, r_sf)
obs.r.dists <- unclass(obs.r.dists)
# Inverse distance interpolation ----
## pred = 1/dist^idp
idp <- 2
inv.w <- (1/(obs.r.dists^idp))
z <- (t(inv.w) %*% matrix(mesh_sf$z)) / apply(inv.w, 2, sum)
# Remove values far from data
dist.min <- apply(obs.r.dists, 2, function(x) min(x, na.rm = TRUE))
z[dist.min > 2] <- min(z, na.rm = TRUE)
# Fill raster with predictions
r.pred <- r
values(r.pred) <- z
# Plot
rasterVis::gplot(r.pred) +
geom_tile(aes(fill = value)) +
scale_fill_gradient2(low = "#d7191c", high = "#1a9641", mid = "#ffffbf",
midpoint = mean(mesh_sf$z))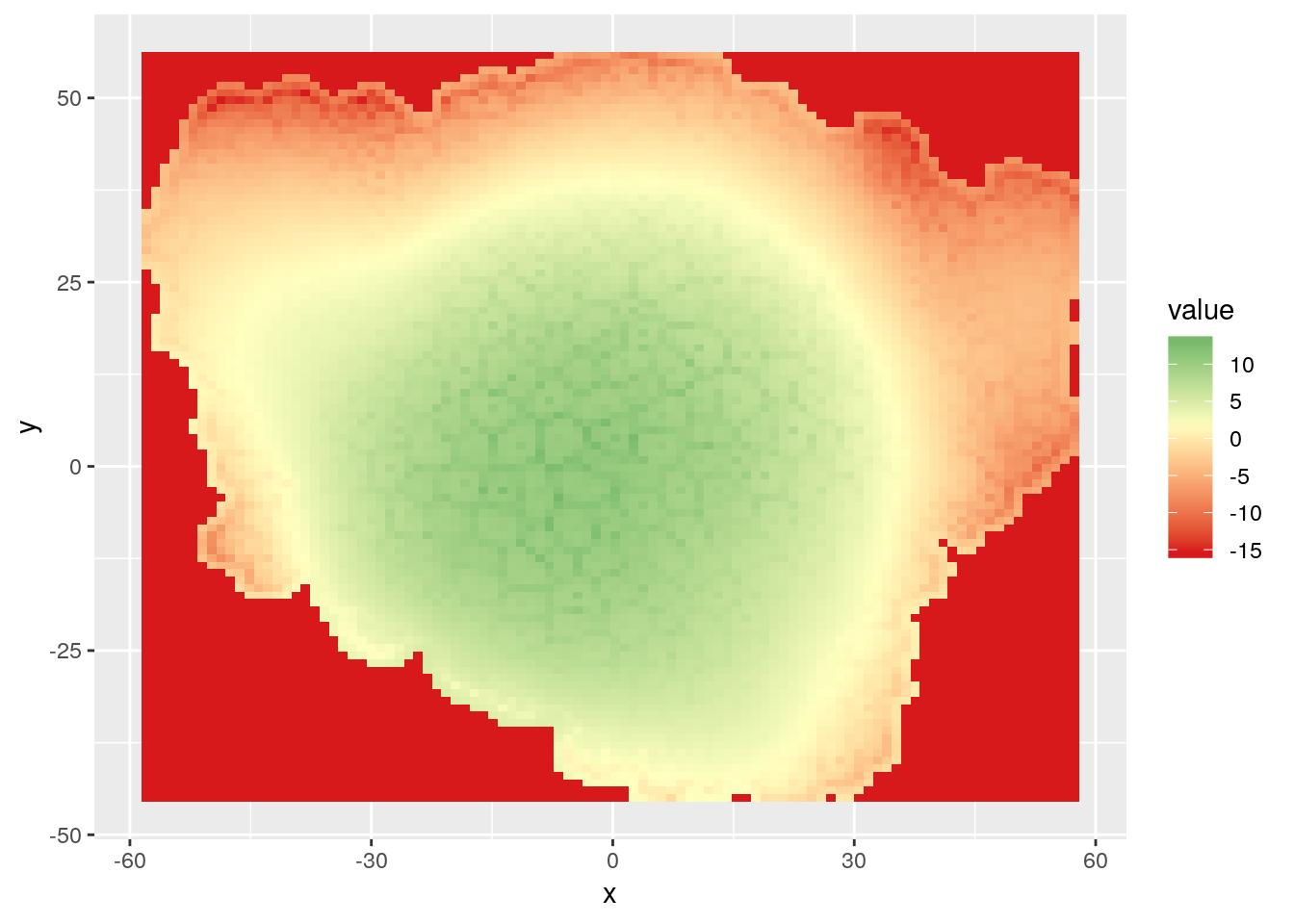
Inclure le raster dans {rayshader}
Et maintenant, jouons avec {rayshader} ! Pour ajouter les points du mesh d’origine sur la vue 3D de {rayshader}, j’ai dû regarder le package de Neil Charles, {geoviz}, qui ajoute des traces GPS à {rayshader}. Ça m’a aidé à trouver comment utiliser et modifier les coordonnées x, y, z de la sortie 3D.
Cette image est un widget rgl interactif
# Transform raster as matrix for rayshader
r.matrix <- matrix(
raster::extract(r.pred, raster::extent(r.pred)),
nrow = ncol(r.pred),
ncol = nrow(r.pred))
zscale <- 1
# Transform points in the scale of matrix in rayshader
mesh_sf_scale <- data.frame(st_coordinates(mesh_sf), mesh_sf$z) %>%
as_tibble() %>%
rename(z = mesh_sf.z) %>%
mutate(X_ray = (X - xmin(r.pred)) / xres(r.pred),
Y_ray = -1*(Y - ymin(r.pred)) / yres(r.pred),
Z_ray = z*zscale + 1.5)
# Create shades
try(rgl.close(), silent = TRUE)
r.matrix %>%
sphere_shade(zscale = 0.1, texture = "unicorn", progbar = FALSE) %>%
add_shadow(ray_shade(r.matrix, zscale = 500, progbar = FALSE), 0.5) %>%
plot_3d(r.matrix, zscale = zscale, theta = -45, zoom = 0.5, phi = 30)
# Add mesh
spheres3d(mesh_sf_scale[,c("X_ray", "Z_ray", "Y_ray")],
col = "grey40", radius = 0.2, add = TRUE)
# Change view
view3d(theta = 30, phi = 45, zoom = 0.5)
# Output html widget for Rmd
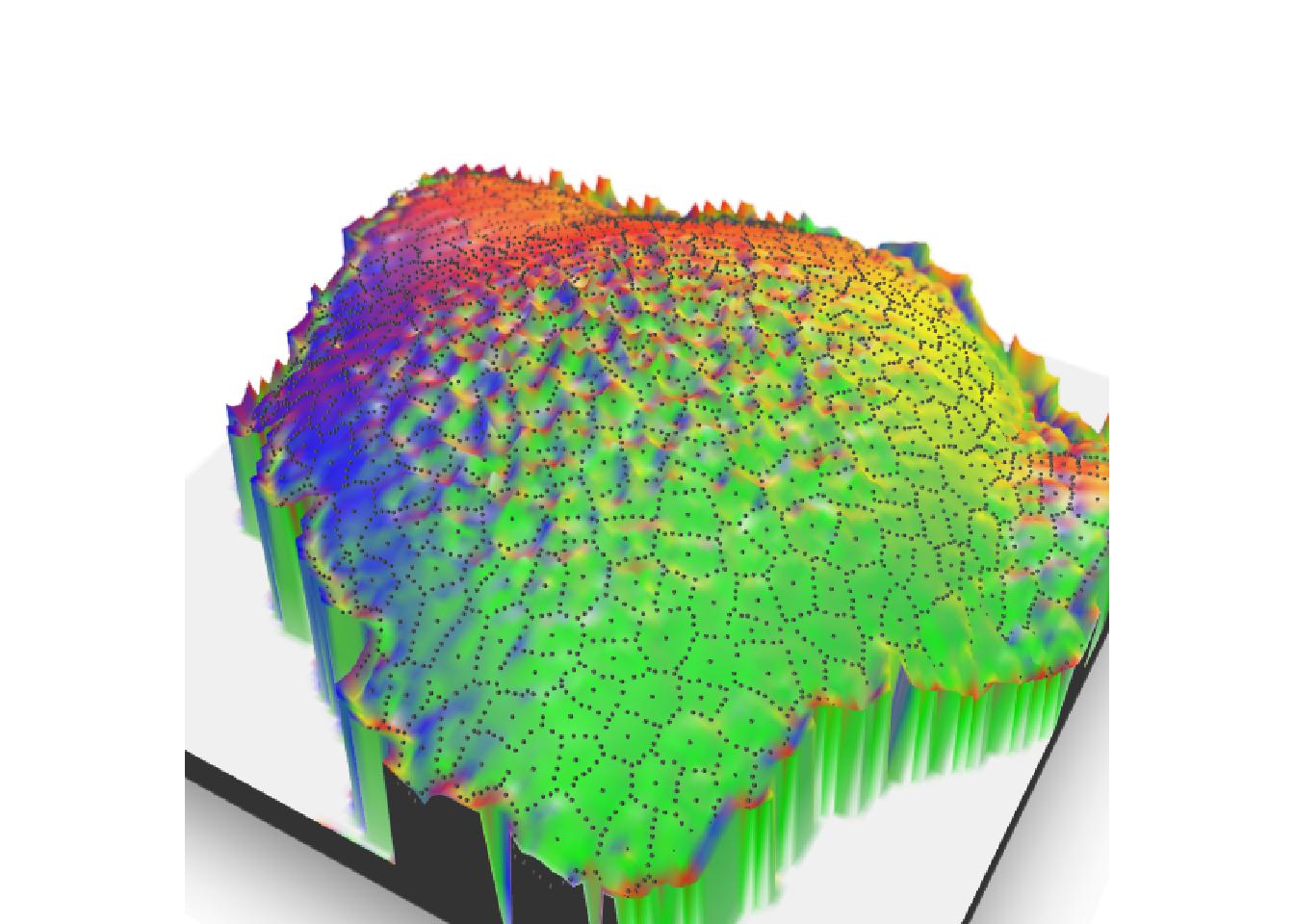
rglwidget()Ajoutez le flou du champ de vision avec {rayfocus}
Il n’y a pas besoin de transformer la sortie 3D pour ajouter le flou sur votre image. La fonction render_depth de {rayshader} récupère les informations nécessaires et utilise {rayfocus} pour créer l’image souhaitée.
render_depth(
focus = 0.5, focallength = 10,
bokehshape = "hex", bokehintensity = 5,
progbar = FALSE
)
Finir avec un GIF
On ne va pas s’arrêter là après tous ces efforts ! Ajoutons encore une petite touche de fun avec un gif en utilisant {magick} !
seq_focus <- seq(0.4, 1, length.out = 35)
for (i in 1:length(seq_focus)) {
view3d(theta = 30, phi = 35 + i/2, zoom = 0.5)
render_depth(
focus = seq_focus[i], focallength = 25,
bokehshape = "hex", bokehintensity = 5,
progbar = FALSE,
filename = file.path(extraWD, "gif",
paste0(formatC(i, flag = "0", width = 3)))
)
if (i == 1) {
all_img <- image_read(file.path(extraWD, "gif",
paste0(formatC(i, flag = "0", width = 3), ".png")))
} else {
new_img <- image_read(file.path(extraWD, "gif",
paste0(formatC(i, flag = "0", width = 3), ".png")))
all_img <- c(all_img, new_img)
}
}
# Reduce width of images
gif <- image_animate(all_img, fps = 5, loop = 0) %>%
image_apply(FUN = function(x)
image_resize(x, geometry = geometry_size_pixels(width = 500)))
# Save as gif in external directory
image_write_gif(gif, path = glue("{extraWD}/focus-1.gif"),
delay = 0.25)
# Reduce size on disk
system(glue("convert {extraWD}/focus-1.gif -fuzz 10% -layers Optimize ../../static{StaticImgWD}/focus-low-1.gif"))
Et voilà ! J’espère que cet article de blog aide à ouvrir votre esprit sur les possibilités offertes par les outils de cartographie, même si ce n’est pas pour faire de la carto !
Le script R complet de cet article de blog se trouve sur mon dépôt Github “Blog Tips”.
Citation :
Merci de citer ce travail avec :
Rochette Sébastien. (2018, oct.. 28). "Jouer avec des outils de cartographie sur des images 3D de cellules". Retrieved from https://statnmap.com/fr/2018-10-28-jouer-avec-des-outils-de-cartographie-sur-des-images-3d-de-cellules/.
Citation BibTex :
@misc{Roche2018Jouer,
author = {Rochette Sébastien},
title = {Jouer avec des outils de cartographie sur des images 3D de cellules},
url = {https://statnmap.com/fr/2018-10-28-jouer-avec-des-outils-de-cartographie-sur-des-images-3d-de-cellules/},
year = {2018}
}

